Soyut
Yüzey antijenik varyasyonu, insanları enfekte eden başlıca patojenler için hayati önem taşır. Bağışıklık sisteminden kaçmak için çeşitli mekanizmalardan yararlanırlar. Bu mekanizmaları anlamak, neden oldukları ölümcül hastalıkları daha iyi önlemek ve onlarla savaşmak için önemlidir. Bağışıklık sistemi zayıflamış bireylerde yaşamı tehdit eden zatürreye neden olan mantar Pneumocystis jirovecii tarafından kullanılanlar hala yeterince anlaşılmamıştır.
Burada, bu mantar şu anda yetiştirilebilir olmasa da, subtelomerik dizi motiflerinin ve yüzey proteinlerini kodlayan genlerin ayrıntılı analizimiz, sistemin her suşta bulunan yaklaşık 80 ifade edilmeyen genin repertuarının yeniden düzenlenmesini içerdiğini ve bunlardan karşılıklı olarak özel ifade için tek genlerin alındığını göstermektedir. Yeni repertuarların, sözde sağlıklı taşıyıcı bireyler tarafından dağıtılması, farklı ülkelerden hastalarda aynı aleller gözlemlendiği için çok verimli görünmektedir. Gözlemlerimiz, antijenik varyasyonun benzersiz bir stratejisini ortaya koymaktadır. Ayrıca, DNA tripleksleri oluşturan küçük kusurlu ayna dizilerinin genom yeniden düzenlemelerindeki olası rolü de vurgulamaktadırlar.
Devamını oku: Mozaikçilik ve subtelomerik gen repertuarlarının yeniden düzenlenmesi kullanılarak mantar antijenik varyasyonugiriiş
Mantar Pneumocystis jirovecii, özellikle insan akciğerlerini kolonize eden zorunlu bir biyotrofik parazittir 1. Bağışıklık sistemi baskılanmış hastalarda, çoğunlukla HIV pozitif hastalarda ve transplant alıcılarında, en sık görülen invaziv fungal enfeksiyonlardan biri olan yaşamı tehdit eden bir pnömoniye neden olur 2. Şu anda, uzun vadeli bir in vitro kültür yönteminin olmaması nedeniyle P. jirovecii biyolojisinin incelenmesi zordur 3 .
Bu mantar, kolonizasyon sırasında konakçının bağışıklık tepkilerinden kaçmaya yardımcı olabilecek mantar duvarlarında yaygın olarak bulunan kitin, α-glukanlar ve dış zincir N-mannanlardan yoksundur 4 . Ayrıca, insanlar için patojenik olan diğer önemli mikroplar gibi, hayatta kalmak için elzem görünen bir yüzey antijenik varyasyon sistemine sahiptir çünkü oldukça sıkıştırılmış genomunun %6’sına kadarını temsil eder 5 , 6 , 7 . Bu sistemin en önemli oyuncusu, altı ana yüzey glikoprotein ailesini (Msg-I ila VI 4 , 8 ) içeren bir süper ailedir. Bunların akciğerlerde ve makrofajlarda bulunan çeşitli insan proteinlerine yapışmaktan sorumlu olduğu varsayılmaktadır 9 , 10 , 11 , 12 . Tüm Msg’lerin, yalnızca askosporların yüzeyinde bulunabilen aile VI hariç, çoğalma sırasında mevcut olan asci ve trofik hücreleri kapsadığına inanılmaktadır; Pneumocystis murina’da olduğu gibi, fareleri spesifik olarak enfekte eden 13. Aile I, hücre yüzeyindeki gen, transkript ve protein sayısı bakımından en bol olanıdır 4 , 14 , 15 . Zatürre sırasında, aile I ve III sırasıyla msg transkriptlerinin yaklaşık %85 ve %10’unu temsil ederken , diğer ailelerin her biri yalnızca %1’dir 15 . Msg’leri kodlayan genler, P. jirovecii’nin 17-20 kromozomlarının tüm alt telomerlerinde bulunur , aile I’in genleri telomerlere en yakın olanlardır 4 , 8 . Alt telomerik lokalizasyon, telomerlerin meyotik buketi içinde ektopik rekombinasyonları, gen susturulmasını ve muhtemelen mutagenezi destekler 16 . Ek olarak, bu genomik bölgedeki gen rekombinasyonları avantajlıdır çünkü genel kromozom yapısı üzerinde hiçbir etkisi yoktur veya çok az etkisi vardır 6 . II-VI ailelerinin genlerinin her birinin kendi promotörü vardır ve bunlar sürekli ve eş zamanlı olarak ifade edilebilir 15 . V ve VI hariç her ailenin birkaç alelinin tek bir enfeksiyon sırasında ifade edildiğini gözlemledik. Ancak, bu ailelerin her birinin tüm veya sadece bazı genlerinin tek hücrelerde ifade edilip edilmediği bilinmemektedir. Bu düzenleme hücre döngüsü sırasında veya telomerlerin yakınlığı nedeniyle susturularak meydana gelebilir (“telomer pozisyon etkisi”) 16 . Öte yandan, aile I’in yaklaşık 80 geninden birinin, genomda tek kopya promotörünün varlığı sayesinde, sözde yukarı akış korunan dizi (UCS) içinde bir hücrede aynı anda ifade edildiğine inanılmaktadır 17 , 18. UCS’nin sonunda, 33 bps uzunluğunda bir dizi, korunan rekombinasyon bağlantı elemanı (CRJE) bulunur ve bu aynı zamanda tüm msg -I alellerinin her birinin başlangıcında bulunur 19 . Bu, büyük olasılıkla, aşağı akışta ifade edilen alelin değişimine izin veren tercih edilen rekombinasyon bölgesidir. Hücre başına ifade edilen tek msg -I geninin kendiliğinden değişiminin, her biri farklı bir msg -I alelini ifade eden hücre alt popülasyonları yarattığı düşünülmektedir . Antijenik varyasyonun ikinci olası mekanizması, msg gen dizilerinin intragenik rekombinasyonlarına , yani gen mozaikliğine dayanır 4 , 8 , 18 , 20 .
Mevcut çalışma, farklı coğrafi konumlardaki hastalarda bulunan msg -I genlerinin repertuarlarını inceleyerek P. jirovecii’nin antijenik varyasyon sisteminde yer alan mekanizmaları daha iyi anlamayı amaçlamaktadır . Gözlemlerimiz, P. jirovecii’nin yüzey antijenik varyasyon sistemi için bir model önermemize olanak tanır .
Sonuçlar
Msg -I alellerinin çoğaltılması ve tanımlanması
Beş farklı coğrafi konumdan Pneumocystis pnömonisi olan 24 hastanın BAL’larında bulunan msg -I alellerini çoğalttık (Ek Tablo 1 ). İki farklı PCR’de, hem ifade edilen hem de ifade edilmeyen tüm msg -I genlerini (bundan sonra “genomik repertuar” olarak adlandırılacaktır) veya tüm ifade edilen msg -I genlerini (“ifade edilen repertuar”) spesifik olarak çoğaltmak için genel primerler kullanıldı. PCR ürünleri PacBio dairesel konsensüs dizisi (CCS) kullanılarak dizilendi. Okumalar, mevcut farklı biyolojik alellerin tanımlanmasına ve bolluğunun belirlenmesine adanmış özel olarak geliştirilmiş bir biyoenformatik boru hattı kullanılarak işlendi. PCR çoğaltımı sırasında oluşturulan kimerik diziler ve PacBio dizileme hatalarının temsil ettiği bilinen sorunlar özel olarak ele alındı ve aşağıda sunulan sonuçları etkilemediğine inanılmaktadır (bkz. “Yöntemler”). Bununla birlikte, bildirilen alellerin çeşitliliği hafife alınmış olabilir.
Hastalarda tanımlanan msg -I alelleri
24 hastadan alınan 48 PCR ürünü arasında 1007 farklı msg -I aleli tanımlandı. Bunların ortalama çiftler halinde dizi özdeşliği %65,7 ± SD %9 idi. Bu değer, daha önce yayınladığımız İsviçre’den 11 alele dayalı değerle (71% ± SD %7) 8 ve ABD’den 80 msg -I geni koleksiyonundaki %70,5 ± SD %3’lük değerle uyumludur 4 . Psödogenleri en az bir durdurma kodonu olan aleller olarak tanımlarsak, bunlar 1007 alelin %5,6’sını temsil eder. Bu, CRJE’li 28 gen arasında %11’lik önceki tek gözlemle tutarlıdır 8 . Bu nedenle, 1007 alelin %94,4’ü herhangi bir intron olmaksızın tamamen açık bir okuma çerçevesi sundu.
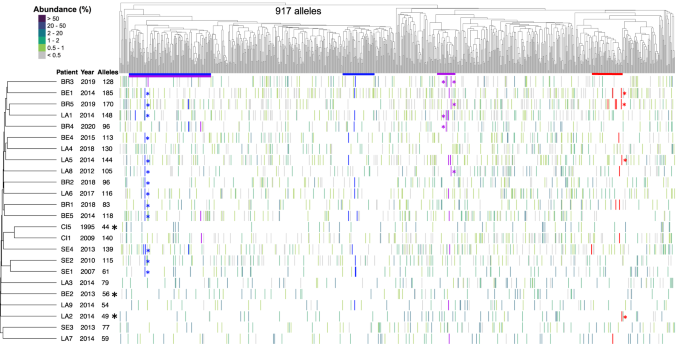
24 genomik ve 24 ifade edilmiş repertuar sırasıyla hiyerarşik sınıflandırma ağaçları kullanılarak sıralanan 917 ve 538 farklı alel içeriyordu (Şekil 1 ). Her iki ağaç da msg -I alellerinin iki ana ve bir küçük alt grubunun varlığını vurguladı ; bu, önceki gözlemlerle tutarlıdır 4 , 8 , 20 . İki ana alt grup, aralarındaki rekombinasyonların oluşumuna dayanarak tek bir aileyi oluşturur 8 , 20 . Daha küçük olanı, henüz aile I’e sınıflandırılamayan aykırı msg genlerine karşılık gelir 8 . Bu alt grupların önemi şimdiye kadar açıklanamamıştır.
Şekil 1: Genomik ve ifade edilen msg -I repertuarlarının bileşimi.
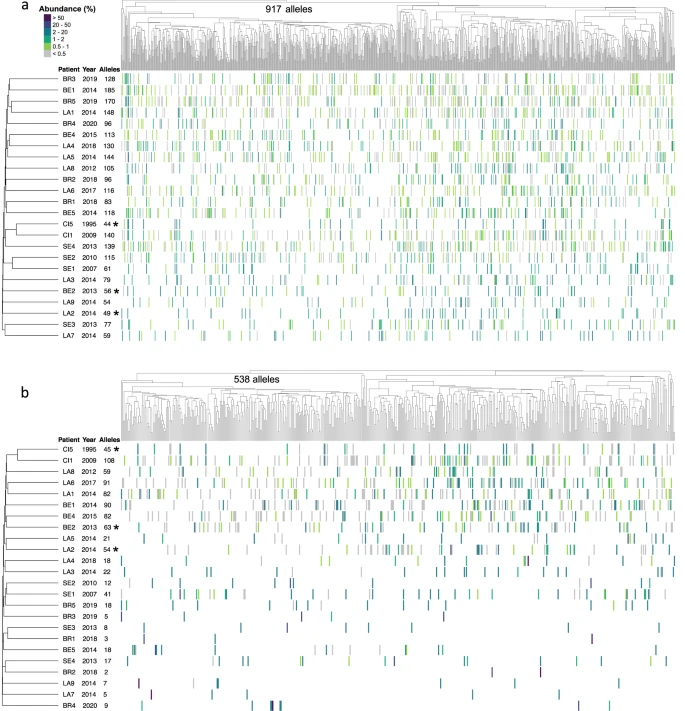
Hastalarda bulunan msg -I alellerinin repertuarları
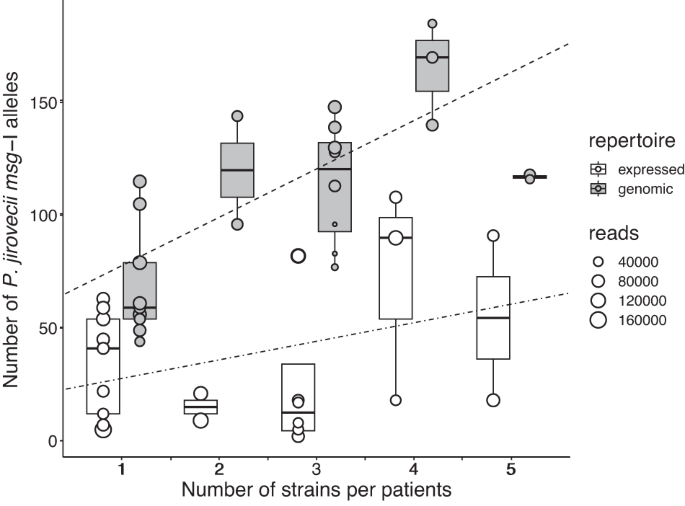
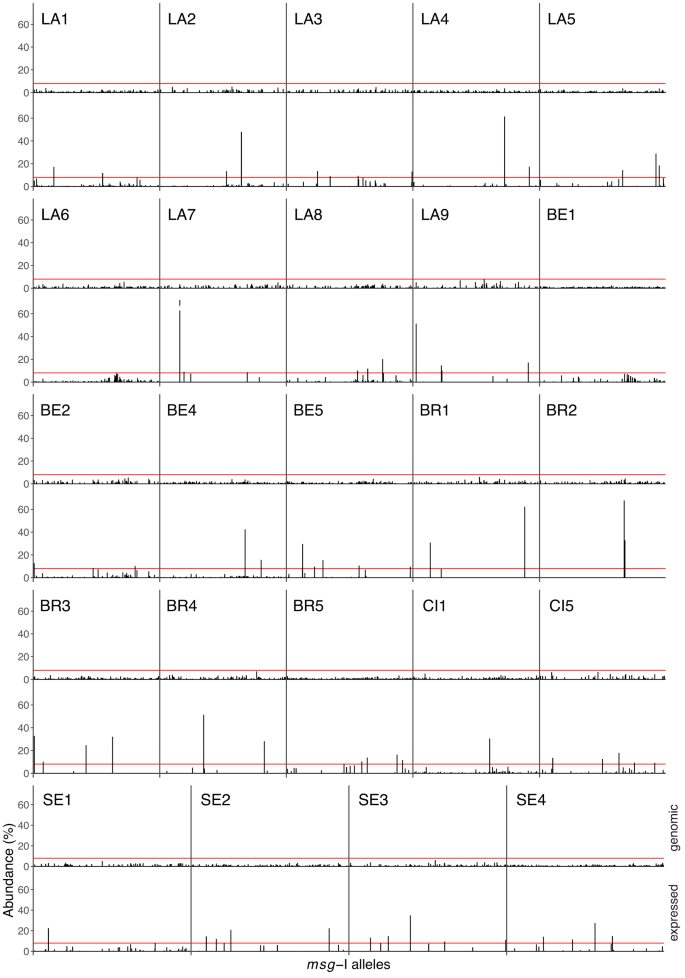
Her hastada bulunan hem ifade edilen hem de genomik repertuarların alelleri, alel ağaçları boyunca düzgün bir şekilde yayılmıştı (Şekil 1 ). Belirli repertuarlarla ilişkili msg -I dizilerinin kladları yoktu ; bu da örnek kökeninin ağaç topolojisini açıklamadığını gösteriyordu. Genomik repertuarlar hasta başına 44-185 alel içeriyordu (104 ± SD 40), buna karşın ifade edilen repertuarlarda 2-108 alel mevcuttu (37 ± SD 34, Ek Tablo 5 ). Genomik ve ifade edilen repertuarların alel sayısı, örneklerde bulunan mantar yükleriyle önemli ölçüde ilişkili değildi (sırasıyla Pearson korelasyonu 0,15 ve 0,39, p değeri = 0,50 ve 0,08, n = 22; Ek Tablolar 1 ve 5’teki veriler ). Özellikle, Brest’ten alınan beş örnekten üçü (BR1, BR2, BR3) 2, 3 ve 5 alel içeren en az çeşitliliğe sahip repertuvarları barındırıyordu, ancak mantar yükü veya altta yatan hastalıkla herhangi bir korelasyon gözlenmedi.
Genomik repertuarların alel sayısındaki değişim, en azından kısmen hastalarda bulunan P. jirovecii suşlarının sayısıyla açıklanmıştır . Gerçekten de, bu sayılar arasında önemli bir korelasyon gözlemlenmiştir (Şekil 2 ). Öte yandan, ifade edilen repertuarlar hiçbir korelasyon göstermemiştir ve bu da diğer daha önemli parametrelerin dahil olduğunu düşündürmektedir. Bir tekrarlanabilirlik deneyi, tek bir suşla enfekte olmuş hasta LA2’nin barındırdığı genomik repertuarın alellerinin değişen bolluğunun, ikili analizlerde tekrarlanabilir olduğunu göstermiştir (Ek not 1 , Ek Tablo 6 , Ek Şekil 4 ). Bu beklenmedik bir durumdu çünkü bu aleller genom başına tek bir kopya halinde mevcuttu ve dolayısıyla DNA örneğinde eşit bolluktaydı. Bu, (i) alellerin amplifikasyonunun ve/veya PacBio diziliminin değişken verimliliğinden, bazılarının hiç amplifiye edilememesinden ve/veya (ii) düşük miktarda bulunan tespit edilemeyen bir eş-enfekte edici suştan kaynaklanabilir. İlk hipotez daha olasıdır çünkü alel sayısının hafife alınması sonuçları daha iyi açıklayacaktır. Gerçekten de, ikinci bir genotipleme yöntemi kullanılarak tek bir suşla enfekte olduğu değerlendirilen hastaların genomik repertuarlarında literatürde bildirilen 80 alelden daha azını barındırdığını açıklar (sırasıyla CI5, LA2, BE2’de 44, 49, 56; Ek Not 2 , Ek Tablo 7 ). Ayrıca, bir yerine iki suş barındırdığı ortaya çıkan diğer altı hastanın dördünün yalnızca 54, 59, 61 ve 79 alel (sırasıyla LA7, LA9, SE1 ve LA3) barındırmasını da açıklar. Önemli olan, bu hafife almanın, varılan sonuçları etkilememesidir; çünkü hiçbiri bu sayıların mutlak değerlerine dayanmamaktadır.
İki PCR’nin tasarımı nedeniyle, ifade edilen repertuar her hasta için genomik repertuarın bir alt kümesi olmalıdır. Tutarlı bir şekilde, ifade edilen repertuarların alellerinin yüksek oranları, karşılık gelen genomik repertuarlarda da mevcuttu (%85 ± SD %17, %40-100, “% genomik olarak ifade edildi”, Ek Tablo 5 ). 24 hastanın 18’i, muhtemelen metodolojinin sınırlamaları nedeniyle %100’den daha düşük bir oran sundu (yukarıya bakın). Tersine ve beklendiği gibi, genomik repertuarların alellerinin daha düşük oranları, karşılık gelen ifade edilen repertuarlarda mevcuttu (%33 ± SD %32, %2 ila %100, “% genomik olarak ifade edildi”, Ek Tablo 5 ). Bu son oranlar, 28 gen arasında %50’lik tek bir önceki tahminle tutarlıdır 8 , bunlar hem ifade edilen hem de ifade edilmeyen alellere karşılık gelir.
Hastalar arasındaki msg -I repertuarlarının benzerliği
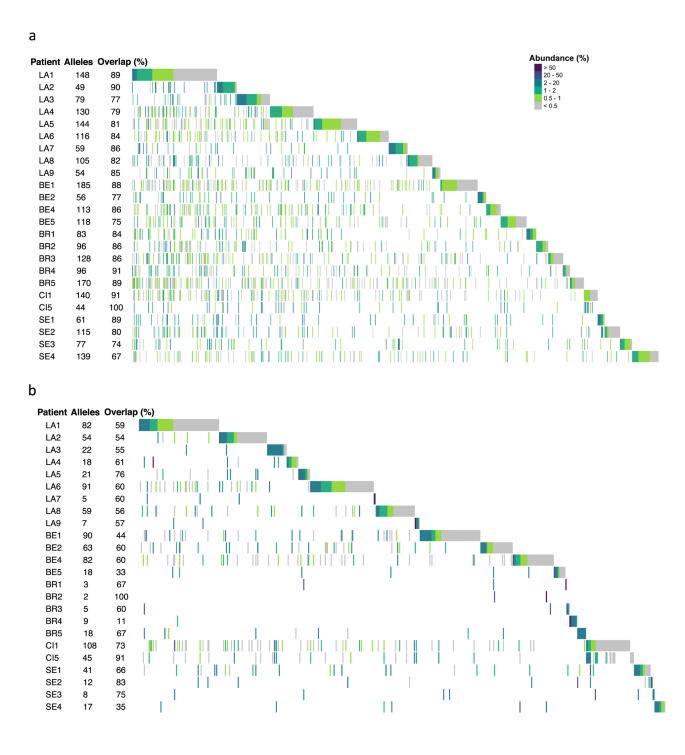
Hastalarda bulunan genomik ve ifade edilen msg -I repertuarları da hiyerarşik sınıflandırma ağaçları kullanılarak sıralandı (Şekil 1 ). Bu ağaçların incelenmesi, her repertuarın diğerlerinden belirgin şekilde farklı olduğunu ortaya koydu. Bu farklılık, repertuarların, örneğin toplandığı yıl veya şehir, hastayı etkileyen altta yatan hastalık veya enfeksiyona neden olan P. jirovecii genotipi(leri) ile belirgin bir korelasyonunun olmadığını ima ediyor. Seville’den alınan iki örneğin genomik repertuarları, SE1 ve SE2 ile Cincinnati’den alınan iki örneğin genomik ve ifade edilen repertuarları, CI1 ve CI5, hafifçe ilişkili oldukları için olası istisnalardı.
Hepsi farklı olsa da, repertuarlar birçok aleli paylaşıyordu. Gerçekten de, her genomik repertuarın alellerinin %84 ± SD %7’si başka bir hastanın en az bir genomik repertuarında mevcuttu. Dikkat çekici olan, bu paylaşım düzeyinin dizilerin kırpılması ve genomik repertuarlarda bulunan alel sayısının olası düşük tahmini nedeniyle kesin olmayabileceğidir (önceki bölüme bakın). Ancak, bu durum çıkarılan sonuçları etkilemez çünkü bunlar mutlak değerlere dayanmamaktadır. İfade edilen repertuarlar için değer %61 ± SD %19’du. Şekil 3, her repertuar tarafından paylaşılan alellerin oranını verir ve repertuarların örtüşmelerinin şehir ve kıtaya ve ayrıca toplama yılına göre önemli ölçüde farklılık göstermediğini görselleştirmeye olanak tanır.
Şekil 3: Şehirlere göre sıralanmış 24 hastanın genomik ve ifade edilmiş repertuarları.
Hastalar arasında msg -I alellerinin dağılımı
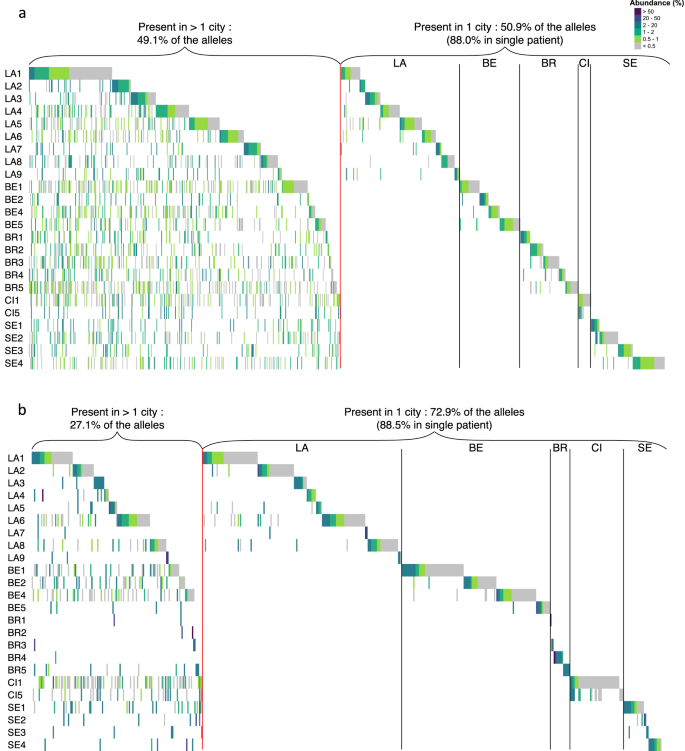
24 genomik repertuarda bulunan 917 alelin yaklaşık yarısı sadece bir şehirde bulundu ve bunların büyük çoğunluğu tek bir hastada bulundu (%88,0, 467 arasında 411, Ek Veri 6’daki Tablo 1 ). Dolayısıyla, 24 hastanın her biri sadece bir hastada gözlemlenen 411 alelden birkaçını barındırıyordu. Kalan yarısı birden fazla şehirde mevcuttu ve %3,6’sı beş şehrin hepsinde de bulunuyordu. 24 ifade edilen repertuarda sadece bir şehirde bulunan alellerin oranı genomik repertuarlardakinden daha yüksekti (%72,9), ancak benzer şekilde yüksek bir oran tek bir hastada bulundu (%88,5, 392 arasında 347, Ek Veri 6’daki Tablo 1 ). Beş şehrin hepsinde tek bir ifade edilen alel mevcuttu (%0,2). Şekil 4 bu oranların görselleştirilmesine olanak sağlıyor ve beş şehrin her birinde karşılaştırılabilir sonuçların gözlemlendiğini gösteriyor.
Şekil 4: Şehirlere göre sıralanmış 24 hastanın genomik ve ifade edilmiş repertuarları ve alellerin bir veya daha fazla şehirdeki varlığı.
Hastalar arasındaki alel dağılımlarını etkileyen parametreleri anlamak için, silico olarak iki simülasyon deneyi gerçekleştirdik. İlk olarak, alellerin alındığı rezervuarın boyutunu araştırdık. 24 genomik repertuvarı, 1000-5000 alel içeren simüle edilmiş bir rezervuardan, ortalama olarak içlerinde bulunan 104 alelin 24 katını çekerek simüle ettik. Bu çekimi 30 kez tekrarladık ve ardından her alelin ortalama çekim sayısını belirledik. 2000 alel içeren rezervuarla elde edilen dağılım, verilerimizin genomik repertuarlarında gözlemlenen dağılıma en çok benziyordu (Ek Şekil 5a ). Benzer şekilde, ifade edilen 24 repertuvarı simüle etmek için 37 alel 24 kez çizildiğinde, 1000 alellik bir rezervuarla elde edilen dağılım, ifade edilen repertuarlar için gözlemlediğimiz dağılıma benziyordu (Ek Şekil 5b ). İkinci simülasyon deneyinde, 24’ten az hastayı analiz etmenin etkisini belirledik. 5, 10, 15 veya 20 hastayı rastgele 30 kez çektik ve gözlemlenen alellerin ortalama sayılarını hesapladık. İlk simülasyon deneyine uygun olarak, alel sayıları düzenli olarak arttı ve 24 hastanın analiziyle tam rezervuara karşılık gelen bir platoya ulaşılmadığını gösterdi (Ek Şekil 6 ). Bu simülasyonlar, tek hastalarda yalnızca bir kez gözlemlediğimiz alellerin yüksek oranının, büyük bir alel rezervuarı ile açıklanabileceğini düşündürmektedir. Ancak, karşılıklı olarak dışlayıcı olmayan bir hipotez, bunların bir kısmının her hastada mozaikçilik tarafından oluşturulan yeni alellere karşılık gelmesidir çünkü bu muhtemelen mantarın hayatta kalması için gereklidir.
Hastalarda msg -I alellerinin bolluğu
P. jirovecii hücrelerinin her popülasyonunun , her biri ayrı bir msg -I aleli ifade eden alt popülasyonlardan oluşması beklenir . Bu alt popülasyonların büyüklüğü ve dolayısıyla ifade edilen alellerin bolluğu, konak bağışıklık sistemine karşı olası bir avantaja veya diğer parametrelere göre değişebilir. Bu hipotezi test etmek için, bir alelin bolluğu, verilen repertuarda bulunan toplam sayının yüzdesi olarak okunma sayısı olarak tanımlandı. Bu bolluklar, ıslak laboratuvarda alt klonlama kullanılarak doğrulandı (Ek Not 3 , Ek Tablo 8 ). 24 genomik repertuarın tümü, maksimum %8’in altında kalan alel bolluğunun yeterince dağılmamış bir dağılımını gösterdi (Şekil 5 ). Buna karşılık, ifade edilen 24 repertuardan 22’si, yani LA6 ve BE1 hariç, bu bollukların aşırı dağılmış bir dağılımını gösterdi; 6’ya kadar çok bollukta alel (≥%8) ve 107’si düşük bollukta alel (<%8) vardı. En yüksek bolluk, %71,9 ile LA7 hastasında görüldü. Bolluk oranı ≥%8 olan 62 ifade edilen alel arasında, 40’ı birden fazla hastada (%64,5) gözlendi. Bu, <%8’de olanlar da dahil olmak üzere tüm ifade edilen aleller arasındaki aynı oranla büyük ölçüde tezat oluşturuyordu (%35,5, %100 – %64,5, Ek Veri 5 ). Bu fark, ≥%8’deki alellerin, örneğin onları barındıran bireylerin konak bağışıklık sistemine göre seçici bir avantaj sağlamış olabileceğini düşündürmektedir.
Şekil 5: Genomik (üst sıra) ve ifade edilen (alt sıra) repertuarlarda bulunan alellerin bolluğu.
İfade edilen repertuar karakterizasyonunun teknik değişkenliği, bu çalışmanın en büyük sınırlamasıdır (Ek Not 1 , Ek Tablo 6 , Ek Şekil 4 ). Bununla birlikte, alel bolluğunun aşırı dağılımı, analiz ettiğimiz dört ifade edilen repertuarın her iki kopyasını da ilgilendiriyordu (Ek Şekil 4b ). Dahası, en az bir kopyada bolluğu ≥%8 olan 11 alelin hepsi her iki kopyada da mevcuttu, bunlardan altısı diğer kopyada <%8’di. Dolayısıyla, yöntemin sınırlaması nedeniyle ifade edilen bollukların değişmesine rağmen, bollukların dağılımları genomik ve ifade edilen repertuarlar arasında açıkça farklılık gösterdi. Bu, karşılıklı olarak dışlayan ifade hipotezini daha da desteklemektedir. Gerçekten de, ifade edilen alellerin her biri belirli bir aleli ifade eden alt popülasyonlara, en sık alellerden en büyüklerine karşılık gelmesi muhtemeldir.
Msg genlerini çevreleyen diziler
msg -I genleri , CDS’lerinden (kodlama dizisi) önce ve sonra yaklaşık 30 bps uzunluğunda kısa korunan diziler, CRJE ve genomik repertuarların amplifikasyonu için primerlerimizi yerleştirdiğimiz durdurma kodonundan sonra yer alan 31 bps sunar. Diğer ailelerin msg CDS’lerini benzer korunan dizilerin çevreleyip çevrelemediğini araştırmak için, daha önce PacBio dizileme teknolojisini kullanarak tek bir suştan bir araya getirdiğimiz 37 alt telomerden yararlandık (Ek Şekiller 7 ve S8 ) 8 . Bu alt telomerler, altı msg ailesinin 113 farklı msg genini taşır. PacBio P. jirovecii genom birleşimine karşı BLASTn analizlerinde sorgu olarak 20 temsili CDS’nin hemen yukarısında veya aşağısında yer alan 200 bps’yi kullandık . I-V ailelerinin genlerinin çoğu, aynı ailenin bir geninin veya psödogeninin CDS’sini çevreleyen diğer dizilerle, çoğunlukla hem yukarı hem de aşağı yönde çok sayıda önemli isabet üretti ( Ek Veri 6’daki Tablo 2 , Ek Veri 1 , 2 , 3 , 4 ve 5 ). İsabetler, montajda bulunan aynı ailenin yukarı veya aşağı yöndeki dizilerinin değişken oranlarını temsil ediyordu (%20-100). Önemlisi, I ve IV aileleri için, bu isabetlerin sorgu ile özdeşlikleri, genlerin kendileri arasındakilerden yaklaşık %10 daha yüksekti (Ek Veri 2 ). Öte yandan, bu özdeşlikler II, III ve V aileleri arasında benzerdi. Özellikle, I ailesi için, psödogenler için genlere göre daha az isabet bulundu (%7-38’e karşı %67-79). II-V aileleri için isabetlerle hizalamaların incelenmesi, aile I’in aksine, CDS’leri çevreleyen herhangi bir korunmuş diziyi tanımlamadı. Bu analizler, msg CDS’lerinin yukarı ve aşağı akış bölgelerinin, aile VI hariç, aynı ailenin genleri arasında sıklıkla benzer olduğunu ve aile I ve IV’ün bölgelerinin yakındaki genlerden daha da korunmuş olduğunu ortaya koydu.
Bu analizler ayrıca, II ve III ailelerinin genlerinin yukarı akış dizilerinin sıklıkla benzer olduğunu, ancak aşağı akış dizilerinin benzer olmadığını ortaya koydu (Ek Veri 6 ). Bu isabetler ve sorguları arasındaki özdeşlikler, yakındaki genler arasındaki özdeşliklerden çok daha yüksekti (Ek Veri 2 , ortalama fark %29,8 ± SD %5,9). Bu isabetler, diğer ailenin yukarı akış dizilerinin önemli bir kısmını içeriyordu (%50 ila %78). Ayrıca, bu iki ailenin 3, 37, 55 ve 8 numaralı genleri diğer aile ile hiçbir benzerlik göstermedi veya daha az benzerlik gösterdi (Ek Tablo 2 ). İkinci genler, subtelomerlerin merkezinde yer alırken, diğerleri uçlarında yer almaktadır (genler no. 7, 25, 34, 53, Ek Şekil 7 ). Ayrıca, II ve III ailelerinin genlerinin 200 bps yukarısındaki benzerliğin CDS içinde yaklaşık 100 bps’ye kadar uzandığı kanıtlandı (Ek Şekil 9 ). Sonuç olarak, msg aileleri II ve III’ün CDS’lerinin yukarı akış bölgeleri genellikle benzerdir ve yakındaki genlerden daha korunmuştur, özellikle subtelomerlerin distal ucunda bulunan genler arasında, ancak aşağı akış bölgeleri için aynı değildir.
Msg genlerinin mozaikliği
Msg genlerinin mozaikliği , yani potansiyel olarak diğer genlerden kaynaklanan parçalardan oluşmuş olması, aynı ailenin üyeleri arasında kesinlikle rekombinasyonların ve çoğaltılmış parçaların tespitine dayanarak önerilmiştir 8 , 20 . Mevcut çalışma sırasında genomik repertuarlarda tanımlanan 917 msg -I alelinin olası mozaikliğini araştırdık . BLASTn algoritmasını kullandık çünkü geniş bir alel kümesinin analizine izin veriyor. BLASTn karşılaştırmasında en yakın isabet oranına sahip rastgele seçilen üç alelin hizalamalarının incelenmesi, her durumda ≥100 bps boyutunda dört ila sekiz çoğaltılmış parça belirledi (toplam 16 parça, bir hizalama Ek Şekil 10’da gösterilmiştir ). Daha sonra 917 alel içindeki her bir parçayı tekrar BLASTn karşılaştırması kullanarak aradık. Parçaların yakından ilişkili aleller arasında, yani ağacın alellerinin aynı alt grubunda korunduğu görülmüştür (Şekil 6 ). Pnömoni bölümünün yılı veya köken şehri ve kıtası ne olursa olsun, birkaç hastada korunmuştur. Dahası, <200 bps olan iki parça alellerin iki farklı alt grubunda mevcuttu (Şekil 6’da mavi ve mor). Morla etiketlenen parçanın tek durumunda, iki alt grup msg -I alellerinin iki ana alt grubuna ait oldukları için çok uzak alellere karşılık geldi (yukarıdaki “hastalarda tanımlanan msg -I alelleri” bölümüne bakın ). Buna karşılık, kırmızıyla gösterilen ≥200 bps parça yalnızca bir alt grupta dağıldı. 16 kopyalanmış parçanın tamamı için kopyalanmış parçaların boyutuna bağlı olarak aynı dağılımlar gözlendi; on tanesi ≥200 bps ve altı tanesi <200 bps idi; sonunculardan ikisi yine de yalnızca tek bir allel alt grubunda mevcuttu.
Her msg -I geninin başlangıcında bulunan CRJE dizisinin yapısı
Her msg -I geninin başlangıcında korunan 33 bps CRJE dizisi muhtemelen ifade edilen alelin değişimine izin veren rekombinasyon bölgesidir. P. jirovecii’ye özgüdür ve onu hedef alabilecek herhangi bir bölgeye özgü rekombinasyon tespit etmedik ( Ek Notlar 5 ve 6 ). CRJE’nin iki önemli özelliğini tespit ettik.
İlk olarak, CRJE’nin her bir ipliği, kusurlu bir ayna tekrarının parçası olan purinler veya pirimidinler açısından zenginleştirilmiştir (Şekil 7a ). Ayna tekrarlarının kopyaları arasında bir AT bp bulunur (pozisyon 14) ve tekrarın sonunda, pozisyon 26-29’da (TTAA) dört AT bp ters tekrarlar olarak düzenlenmiştir. Bir literatüre göre 21 , 22 , 23 , 24 , CRJE’nin özellikleri, kanonik olmayan bir H-DNA’nın iki izomerini, yani *H-DNA’yı oluşturabileceğini göstermektedir (Şekil 7b ). *H-DNA, bu durumda Hoogsteen bağlarını içeren ve 12 bps’lik tek zincirli bir uzantı sunan 11 baz üçlüsünden oluşan bir molekül içi DNA üçlüsüdür (Ek Şekil 12a , b baz üçlülerinin yapısını ve DNA üçlüsünün 3B modelini göstermektedir). Biraz daha küçük olmasına rağmen, CRJE dizisi *H-DNA’yı oluşturduğu bildirilen kanonik dizilere oldukça benzemektedir (Ek Şekil 12c ). Tutarlı bir şekilde, CRJE tarafından potansiyel olarak oluşturulan 11 baz üçlüsünden yedisi, *H-DNA’yı oluşturduğu bildirilen en sık ikisinden biridir (CG*G, üçlüdeki * sembolü Hoogsteen bağlarını ifade eder). 11 üçlüden ikisi şimdiye kadar sadece H-DNA’da rapor edilmiştir (CG*C, GC*G), geriye kalan iki GC*C ise kanonik olmayan ve bir DNA üçlüsüne entegre olmak için daha fazla enerji gerektiren ancak in vitro gözlemlenenlerdir. *H-DNA, H-DNA’dan çok daha az katı dizi gereksinimleri sunar 21 , ayna tekrarı mevcut olmayabilir bile, bu nedenle CRJE dizisinin ve oluşturabileceği alternatif üçlülerin özgüllükleri makuldür. DNA üçlülerinin hücrede bir dizi rol oynadığına inanılmaktadır 22 , 23 .
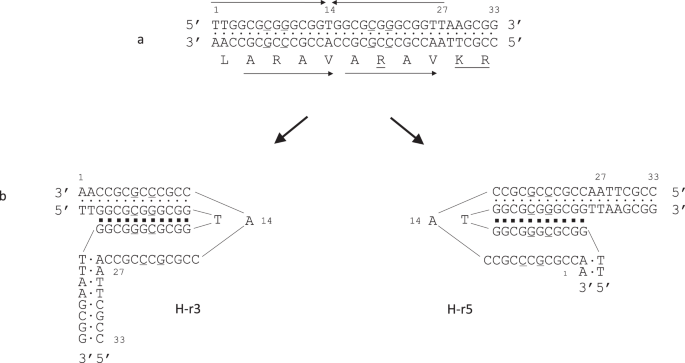
CRJE’nin ikinci yeni özelliği, kodlanan peptidin, UCS 25’e karşılık gelen sabit kısmın çıkarılmasını sağladığına inanılan Kexin’in kesme bölgesinin hemen yukarısında, motif ARAV’ın doğrudan bir tandem tekrarını sunmasıdır . İlginç bir şekilde, tekrarlarda kusurlu olan dört pozisyondan biri, pozisyon 19’daki C, motif ARAV’ın ikinci kopyası içindeki arginini (R) kodlamak için gereklidir (Şekil 7a ). ARAV içindeki RA, katepsinler (MEROPS veritabanı) gibi bir dizi peptidaz tarafından kesilen bir motiftir. Dahası, R kalıntısı tripsin benzeri peptidaz için ortak bir tanıma bölgesidir. Bu nedenle, CRJE dizileri, her bir Msg’nin antijenisitesinin çeşitliliğini sağlamak için enzimler tarafından Msg-I proteinlerinin sabit kısmının uygun şekilde çıkarılmasında da yer alabilir.
Cinsin diğer türleri söz konusu olduğunda, Pneumocystis mac acae ve Pneumocystis oryctolagi, Kexin’in tanıma bölgesinde bulunana ek olarak iki R kalıntısının varlığı da dahil olmak üzere, P. jirovecii’ninkine benzer bir CRJE dizisine sahiptir (Ek Not 7 , Ek Şekil 13 ). Bu nedenle, bu CRJE’ler ayrıca bir DNA üçlüsü oluşturabilir ve başka bir proteaz tarafından tanınabilir. Öte yandan, diğer dokuz Pneumocystis türü, *H-DNA üçlüleri oluşturan kanonik ayna tekrarından daha uzakta bir CRJE barındırır. Ancak, yine de böyle bir yapı oluşturabilirler çünkü *H-DNA üçlüsünün oluşumu, kanonik H-DNA’dan daha çok yönlüdür ve dizi düzeyinde daha az gerektirir 21 .
Tartışma
Mantar P. jirovecii’nin antijenikliğini değiştirmek için kullandığı mekanizmaları , ana yüzey proteinlerini kodlayan genlerin repertuarlarını ve bu genleri barındıran alt telomerlerde bulunan motifleri ve benzerlikleri analiz ederek araştırdık. Yeni sonuçlarımıza dayanarak, P. jirovecii’nin antijenik varyasyon sisteminin önem sırasına göre listelenen aşağıdaki üç mekanizmaya dayandığını varsayıyoruz:
- (Ben)Msg -I gen repertuarlarının yeniden düzenlenmesi ve ifade edilen alelin tüm genlerin translokasyonu yoluyla değiştirilmesi.
- (ii)Tekli rekombinasyonlar yoluyla subtelomerlerin yeniden düzenlenmesi.
- (iii)Gen içi rekombinasyonlar yoluyla msg genlerinin mozaikleşmesi .
Hepsi farklı olsa da, P. jirovecii msg -I alellerinin genomik repertuarları hastalar arasında önemli ölçüde örtüşüyordu. Bu, ifade edilmeyen tüm alellerin çok sık translokasyonlarının subtelomerler arasında meydana geldiği ve bu da alellerin yeni çeşitlerini ve dolayısıyla yeni subtelomerleri yarattığı anlamına gelir. Daha sonra, msg -I alellerinin daha fazla translokasyonunun meydana geldiği farklı P. jirovecii hücre hatlarına ayrılırdı. Alellerin bu sürekli yeniden düzenlenmesi, coğrafi konumdan, Pneumocystis pnömonisi bölümünün yılından veya hiyerarşik sınıflandırmalarıyla gözlemlenen alel alt gruplarından bağımsız olarak gözlemlediğimiz her repertuarın farklılığına yol açardı . Bu translokasyonların olası altta yatan mekanizması, msg -I CDS’nin yukarı akışında yer alan bölgede ve aşağı akış bölgesinde yer alan iki homolog rekombinasyonun birleşimidir (Şekil 8a ). Bu tür olaylar iki alelin değişimine (karşılıklı değişim) izin verir, ancak muhtemelen birinin diğeriyle değiştirilmesine de (dönüşüm, karşılıklı olmayan değişim) izin verir. Tutarlı bir şekilde, msg -I CDS’lerini çevreleyen yukarı ve aşağı akış bölgelerinin, genlerin kendileri arasındakinden yaklaşık %20 daha yüksek bir özdeşlikle benzer olduğu kanıtlandı, bu da muhtemelen genlerin dışındaki varsayılan rekombinasyonları destekledi. Bu rekombinasyonlar, her CDS’nin başlangıcında bulunan 33 bps CRJE dizisinde ve durdurma kodonundan sonra bulunan 31 bps dizisinde tercihen meydana gelebilir, çünkü bu diziler bugüne kadar bildirilen tüm msg -I gen dizilerinde tamamen korunmuştur. Ayrıca, msg aileleri II, III, IV ve V’teki CDS’lerin hemen yukarı ve aşağı akışında bulunan dizilerin de, özellikle subtelomerlerin ucunda bulunan genler arasında sıklıkla benzer olduğunu bulduk. Ayrıca, aile IV’tekiler, aile I’deki gibi, CDS’lerin kendilerinden daha yüksek bir kimlik de sunar. Bu gözlemler, tüm genlerin translokasyonunun bu aileleri de ilgilendirebileceğini düşündürmektedir, ancak şu anda bu hipotezi destekleyen hiçbir veri mevcut değildir. MSG genlerinin translokasyonunun, yan genlerin kendilerinden daha fazla korunan intergenik boşluklar sayesinde kolaylaştırılması, daha önce Pneumocystis carinii’de 27 varsayılmıştır .
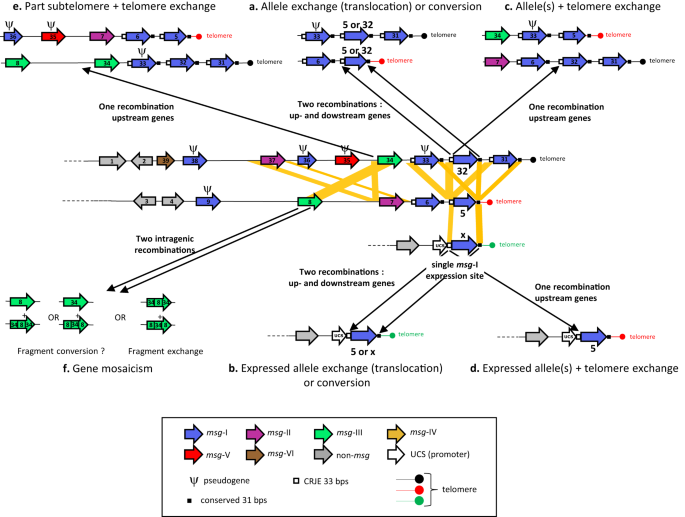
Çift rekombinasyonlar yoluyla gerçekleşen translokasyonların ayrıca ifade edilen msg -I alellerini de etkilemesi muhtemeldir çünkü bunlar CRJE yukarı akışını ve aşağı akışını da sunar (Şekil 8b ). Ancak, çoğu olay muhtemelen ifade edilmeyen alelleri etkiler çünkü genom başına, sonuncular yaklaşık 80 kopya halinde bulunurken, genom başına tek bir ifade edilen alel vardır. Dahası, ifade edilen alelin translokasyonu, yukarı akışındaki benzerliğin yalnızca CRJE yukarı akışının 33 bps’si olması gerçeğiyle azaltılabilirken, ifade edilmeyen genlerinki 200 bps uzunluğunda artı CRJE’nin 33 bps’sidir. Daha önce varsayıldığı gibi 8 , 28 , msg -I genlerinin subtelomerler içindeki distal konumu, tek bir rekombinasyon yoluyla değişimlerini kolaylaştırabileceği için alternatif bir mekanizma önerilmiştir . İkincisi, iki CRJE dizisi arasında meydana gelir ve bu da onlara bağlı telomerlerle birlikte bir veya birkaç genin değiş tokuşuna yol açar (Şekil 8 c, d).
Hasta başına ifade edilen alel sayısı büyük ölçüde değişti (2–108). Mevcut çalışmada kanıtladığımız gibi, hastayı enfekte eden P. jirovecii suşlarının sayısının dışında , bu sayıyı etkileyen aşağıdaki dört parametreyi varsayabiliriz:
- (Ben)Hastayı enfekte eden farklı msg -I repertuarlarına sahip hücrelerin sayısı, başlangıçta ifade edilen farklı alellerin sayısını etkileyebileceğinden.
- (ii)Hastanın daha önce karşılaştığı alelleri veya epitopları ifade eden hücre alt popülasyonlarını az ya da çok etkili bir şekilde ortadan kaldırabilecek düzeydeki immünosüpresyon.
- (iii)Mantarın edinilmesi ile Pneumocystis pnömonisi olayı arasında geçen süre. Gerçekten de, belirli bir aleli ifade eden yeni hücre alt popülasyonlarının sürekli olarak ayrıldığı ve analiz ettiğimiz gibi bağışıklık sistemi baskılanmış hastalarda zamanla birikebileceği varsayılmaktadır.
- (iv)Metodolojinin teknik sınırlamaları, ifade edilen repertuarlarda bulunan genomik repertuarların genel olarak düşük oranında (24 hastanın 13’ünde <%15, Ek Tablo 5 ) rol oynamış olabilir. Bu nedenle, ifade edilen repertuarlardaki alel sayısı hafife alınmış olabilir. Bu, gözlemlerimizin P. carinii ve P. murina enfeksiyonlarında ifade edilen genomik msg -I genlerinin %100’üyle çelişmesini açıklayabilir 4 . Farklı konaklar tarafından uygulanan farklı seçici baskılar da söz konusu olabilir.
CRJE dizisinin subtelomerlerde tamamen ve çoklu kopyalar halinde korunması, P. jirovecii antijenik varyasyon sisteminde önemli bir rol oynadığını güçlü bir şekilde düşündürmektedir . Bu dizilerin potansiyel olarak oluşturabileceği DNA tripleksleri dahil olabilir. Ancak, ayna tekrarlarının ökaryotların genomlarının %1’ine kadarını temsil eden bir ayırt edici özellik oluşturmasına rağmen 29 , 30 , 31 , işlevleri şu ana kadar varsayımsal olarak kalmıştır. Bu durum, DNA triplekslerinin izlenmesinin çok zor olmasından kaynaklanmaktadır 21 , 23 , 32 . Bununla birlikte, çok sayıda dolaylı kanıt, bunların bir dizi genetik süreçte yer aldığını düşündürmektedir 33 . Homolog rekombinasyon, ayna tekrarlarının sıklıkla rekombinasyon bölgelerine yakın olarak bildirilmesi nedeniyle en çok tekrarlanan süreçtir. Aşağıdaki referans listesindeki 28 numaralı referansı silin: 21 , 33 , 34 , 35 , 36 , 37. Ayrıca, nükleik asit tripleksleri (RNA:DNA hibritleri, R-döngüleri) (i) Trypanosoma brucei’nin antijenik varyasyon sistemine katılan ifade edilmiş alelin değiştirilmesinde 38 ve (ii) memeli immünoglobulinlerindeki rekombinasyonların değiştirilmesinde 39 rol oynar. Bu nedenle, CRJE dizileri tarafından potansiyel olarak oluşturulan DNA triplekslerinin, P. jirovecii’nin antijenik varyasyon sisteminde yer alan rekombinasyonları aracılık ettiğini, belki de aktive ettiğini varsayıyoruz .
Mevcut çalışmada, II ve III ailelerinin genlerinin yukarı akış bölgelerinin çok benzer olduğunu, ancak aşağı akış dizilerinin benzer olmadığını bulduk. Kimlikleri, yakındaki genler arasındakinden yaklaşık %30 daha yüksekti (%79,8’e karşı %50,1). Bu benzerlikler, aralarındaki tek bir homolog rekombinasyon yoluyla büyük subtelomer parçalarının değişimini teşvik edebilir (Şekil 8e ). Bu tür genler arası rekombinasyonlar, gen mozaikliğine yol açan gen içi rekombinasyonlar kadar veya daha sık olabilir çünkü her msg ailesindeki genlerin ortalama kimliği daha düşük veya benzerdir, yani sırasıyla I–VI aileleri için 66, 83, 83, 72, 66, 45 8 . Bu mekanizma, potansiyel olarak farklı boyutlarda yeni subtelomerlerin yaratılmasına yol açacaktır. Bu, gözlemlediğimiz telomer uzunluğundaki önemli varyasyonla uyumlu olacaktır 8 (Ek Şekiller 7 ve 8 ). Gerçekten de, CRJE dizisine sahip olan ve telomere her zaman en yakın olan genler olan genomik genler ile msg -I genleri arasındaki mesafe 6 kb (bitişik/alt telomer 149, Ek Şekil 8a ) ile 25 kb (54, Ek Şekil 7 ) arasında değişiyordu.
Subtelomerik genlerle ilgili genel bir fenomen, bunlar arasında, yani homolog olmayan kromozomlar arasında da ektopik rekombinasyonların meydana gelmesidir 16 , 40. Bu rekombinasyonlar çoğunlukla telomerler ve subtelomerler mayozun profazı sırasında iğsi gövdeye bağlanarak bir “buket” halinde demetlendiğinde meydana gelebilir 16 , 41 , 42. Bu tür iki rekombinasyonun ürünlerini değerlendirmek zordur ve organizmaya göre değişebilir (Şekil 8f ). Dahası, tüm organizmalarda parça dönüşümleri ve parça değişimleri arasında bir denge olabilir çünkü ilki genellikle alelleri homojenleştirirken, ikincisi onları çeşitlendirir 43. Dengedeki her bir sürecin önemi, sisteme ithal edilen farklı alellerin miktarına bağlı olabilir, örneğin suşların çiftleşmesiyle, çünkü iki alel setinin kaynaşmasına yol açar. Alellerin mozaikleşmesinden sorumlu rekombinasyonlar, aynı zamanda, parça dönüşümleri yoluyla değişmiş genlerin meiotik onarımından da kaynaklanabilir 44 .
Ektopik rekombinasyonların , gözlemlenen P. jirovecii genlerinin, psödogenlerinin ve subtelomerlerin intergenik boşluklarının mozaiğini oluşturması muhtemeldir . Ancak, mevcut gözlemlerimiz, P. jirovecii msg -I alelleri arasında gözlemlediğimiz kopyalanmış parçaların, alellerin çeşitlenmesi sırasında ata alellerinden korunduğunu göstermektedir. Gerçekten de, incelediğimiz <200 bps parçaların üçte ikisi (6’dan 4’ü), uzak alellerin iki alt grubunda mevcuttu. Biri , msg -I alellerinin iki ana alt grubunda, yani çok uzak olanlarda bile mevcuttu. Öte yandan, ≥200 bps olan on parçanın hepsi tek bir alt grupta mevcuttu. Çeşitlenme süreci sırasında, bir parçanın bölünme olasılığı, <200 olanlara göre ≥200 uzunluğunda muhtemelen daha fazladır. Bu nedenle, daha büyük parçaların daha kısa olanlara göre tek bir alt grupta mevcut olma olasılığı daha yüksektir.
Alternatif bir hipotez, çoğaltılmış parçaların, ilgili alellerin alt grupları içinde gerçekleşen dönüşüm olaylarından ve ardından hastalar arasında oluşturulan alellerin yayılmasından kaynaklandığıdır. Gözlemlediğimiz gibi, iki uzak alt grupta aynı çoğaltılmış parçaların varlığı, aynı çoğaltılmış parçalara yol açan gen boyunca rekombinasyonların sıcak noktalarının varlığıyla açıklanabilir. Ancak, çoğaltılmış parçaların konumlarına ilişkin analizlerimiz, böyle sıcak noktaların varlığını ortaya koymadı. Bu nedenle, gözlemlerimiz, P. jirovecii’deki msg -I alellerinin çeşitlenmesinin , öncelikle ata alellerini bölen rekombinasyonlardan kaynaklandığı hipotezini desteklemektedir. Bu rekombinasyonlar, parça değişimleriyle sonuçlananlar, yaklaşımımızla tespit edemediğimiz olaylar veya yukarıda belirtilen subtelomerlerin yeniden düzenlenmesiyle sonuçlanan tek olanlar olabilir. Bu sonuç, önceki çalışmaların sonuçlarıyla çelişmektedir. Gerçekten de, P. jirovecii msg -I genlerindeki yüksek G + C motif içeriği 45 ve P. carinii ekspresyon bölgesindeki çoğaltılmış parçaların analizi 46 dönüşüm olaylarını önermiştir .
Burada analiz edilen bağışıklık sistemi baskılanmış hastaların akciğerlerinde birçok farklı P. jirovecii msg -I alelinin ifade edildiği gözlemlendi. Buna karşılık, mantar tarafından kolonize edilmiş bağışıklık sistemi yeterli bireyler, etkili bağışıklık sistemleri nedeniyle daha az sayıda ifade edilmiş alel barındırabilir, ancak şu anda hiçbir veri mevcut değildir. İkinci bağışıklık yeterli bireyler muhtemelen P. jirovecii’nin antijenik varyasyon sisteminin evrimleştiği nişi oluşturur, bu nedenle bu sistem her şeyden önce bir kolonizasyon faktörüdür. Bu nedenle, ikincisini daha iyi anlamak için, Pneumocystis pnömonisi olan hastalarla temas halinde olan sağlık çalışanları veya P. jirovecii tarafından ilk enfeksiyonlarını yaşayan bebekler gibi bağışıklık yeterli geçici taşıyıcıları araştırmak yararlı olabilir .
msg -I genlerinin repertuarlarının önemli örtüşmesi, P. jirovecii popülasyonları arasındaki sık temaslar yoluyla alellerin ve suşların yayılması için çok etkili bir aracın varlığını ima eder . Bu, genel popülasyonda sık görülen olaylar olan bebeklerin ilk enfeksiyonları yoluyla gerçekleşebilir. Birbirini dışlamayan bir diğer hipotez ise, sağlıklı insanlar tarafından mantarın geçici olarak taşınmasıdır; bu fenomen sıklıkla hipotez olarak ortaya atılmış ancak şu ana kadar kesin olarak kanıtlanmamıştır 47 , 48 , 49 .
Verilerimiz, P. jirovecii stratejisinin, daha önce önerdiğimiz gibi antijenik olarak farklı yeni alt popülasyonların sürekli üretimi olduğunu desteklemektedir 8 . Bu strateji rekombinasyonlara dayanır ancak bunların sıklığı ve subtelomerlerin zaman içindeki evrim hızı, muhtemelen aynı hastalardan alınan ardışık örneklerin analiziyle belirlenmeyi beklemektedir. Bu tür örneklerin analizi, çalışmamızın incelemediği bağışıklık kaçınmasının önemli bir yönünü ele almamızı da sağlayabilir: hücre yüzeyindeki Msg glikoproteinlerinin zamanla değişimi. Her nesildeki yüzey antijenik varyasyonu muhtemelen hayatta kalmak için gerekli olduğundan, bu mantarın zorunlu cinselliğini açıklayabilir 50 , 51 . Tutarlı bir şekilde, ikincisi muhtemelen mantarın çoğalmasını da sağlar 50 , 51 . P. jirovecii’nin stratejisi, insan patojenleri arasında türünün tek örneğidir ve memeli akciğerlerinin oluşturduğu steril olmayan nişe uyarlanabilir 8 , 52 . Bu, Plasmodium ve Trypanosoma’nın antijenik olarak homojen olan ve ifade edilen genin değişimi üzerine zamanla değişen popülasyonlarıyla çelişir 53 . Bunlar kan gibi steril nişler tarafından empoze edilebilir.
Yöntemler
Etik onay ve katılıma izin
Lozan, Bern ve Sevilla’da hastaneye kabul prosedürü tüm hastalar için bilgilendirilmiş yazılı onam içeriyordu. Kabul formu, örneklerinin araştırma için kullanılmamasını talep etme olasılığını içeriyordu. Örnekler hastanenin rutin prosedürü aracılığıyla elde edildi ve anonimleştirildi. Araştırma protokolü Sevilla Hastanesi inceleme kurulu ve İsviçre kurumsal inceleme kurulu (Commission Cantonale d’Éthique de la Recherche sur l’Être Humain, http://www.swissethics.ch ) tarafından onaylandı. Arşiv örneklerinin toplanması ve kullanımı Brest Üniversitesi Hastanesi etik komitesi tarafından 24 Haziran 2021’de onaylandı ve Fransız Araştırma Bakanlığı ve Agence Régionale de l’Hospitalization’a DC-2008-214 No. ile kaydedildi. Cincinnati’den alınan örnekler anonimleştirildi; kullanımları insan denekler üzerinde araştırma amaçlı değildi ve Kurumsal İnceleme Kurulu’ndan onay gerektirmiyordu.
Örnekler ve DNA çıkarımı
Bronko-alveolar lavaj (BAL) örnekleri , 25 yıl boyunca beş farklı coğrafi konumda Pneumocystis pnömonisi olan 24 immün sistemi baskılanmış hastadan toplandı (Ek Tablo 1 ). Her BAL’ın 0,2 ml’sinden QIAamp DNA Kan Mini Kiti (Qiagen) kullanılarak DNA çıkarıldı.
msg -I genlerinin repertuarlarının güçlendirilmesi
İfade edilen tüm msg -I genlerinin tamamını (“ifade edilen repertuar”) veya örnekte bulunan genomun tüm msg -I genlerinin tamamını, yani ifade edilenler artı ifade edilmeyen genleri (“genomik repertuar”) çoğaltmak için iki ayrı genel PCR kullanıldı. İfade edilen repertuar, CRJE’nin 27 bps yukarısında, UCS dizisinin sonunda bulunan ileri bir primer sayesinde özel olarak çoğaltıldı (GK135: 5′ GACAAGGATGTTGCTTTTGAT 3′) 54 . Genomik repertuar, CRJE dizisinin 3′ yarısını kaplayan ileri bir primer kullanılarak özel olarak çoğaltıldı (CRJE-for-bis: 5′ TGGGCGCGGGCGGT Y AAG 3′; altı çizili Y, CRJE’lerin yaklaşık %89’unun ve %11’inin sırasıyla bu pozisyonda T ve C’yi barındırması nedeniyle tanıtıldı ; Kaynak veriler Kaynak Veri dosyasında sağlanmıştır). Ters primer her iki PCR için de aynıydı ve msg -I genlerinin durdurma kodonundan yaklaşık 90 bps sonra 31 bps’lik korunmuş bir bölgede yer alıyordu (GK452: 5′ AATGCACTTTCMATTGATGCT 3′; altı çizili M, dizilerin yaklaşık %90’ının ve %10’unun sırasıyla bu pozisyonda T ve G’yi barındırması nedeniyle tanıtıldı; bu primer daha önce yayınlanan 20 ile aynıdır , titreme dahil). Bölgeyi de içeren 48 yayınlanmış dizinin hizalanması Ek Şekil 1’de gösterilmiştir , primerin 3′ ucunda bir A yerine bir G’nin varlığı nedeniyle yalnızca biri daha az verimli bir şekilde çoğaltılabilir. PCR, 0,2 µl polimeraz (KAPA LongRange HotStart, Roche) içeren 20 µl’lik son hacimde BAL’dan alınan bir mikrolitre DNA, sağlanan tampon, 0,2 mM’lik her dNTP, 0,5 µM’lik her primer ve 3 mM’lik son bir MgCl2 konsantrasyonu ile gerçekleştirildi . Kontaminasyonları önlemek için PCR’ler her odaya ayrılmış malzemeler kullanılarak fiziksel olarak ayrı odalarda kuruldu ve analiz edildi ve her deneyde sistematik olarak negatif kontroller gerçekleştirildi.
Her iki PCR için de bir touchdown PCR prosedürü kullanıldı. İfade edilen repertuvarı çoğaltmak için program, 62 °C’den 55 °C’ye tavlama sıcaklığının sabit bir şekilde azaltılmasından oluşan 10 döngü ile başladı, ardından 55 °C’de tavlama ile 25 veya yeterli miktarda PCR ürünü elde etmek gerekirse 30 döngü geldi. Genomik repertuar için, azalma 10 döngüde 68 °C’den 58 °C’ye oldu, ardından 58 °C’de tavlama ile 20 veya 25 döngü geldi. Uzama süresi her iki PCR için de 72 °C’de 3 dakikaydı. Reaksiyonlar 72 °C’de 7 dakikalık son bir uzama ile sona erdi. PCR ürünleri (i) agar jel üzerinde yaklaşık 3100 bps boyutlarında olmaları ve (ii) bunlardan bazılarının TOPO klonlama kiti (Invitrogen) kullanılarak alt klonlanması ve ardından Sanger dizilenmesi ile doğrulandı. PCR ürünleri daha sonra, dizileme sırasında bilinmeyen bileşimi nedeniyle herhangi bir sorun yaşanmaması için sağlanan yükleme tamponu yerine %50 gliserol kullanılması dışında, üreticinin talimatlarına göre E-gel CloneWell 0.8% (Invitrogen) ile saflaştırıldı. Son olarak, PacBio dairesel konsensüs dizilemesi için gereken yeterli konsantrasyona ulaşmak üzere tüp kapağı açıkken 50 °C’de bir ısıtma bloğunda buharlaştırıldılar. Her açık tüp, çapraz kontaminasyonu önlemek için buharlaştırma sırasında hava geçirmez olmayan bir kapakla kapatıldı. İkincisinin yokluğu, dizileme sırasında plazmit kontrollerinde tek bir alelin varlığıyla değerlendirildi. 48 numuneden 38’inden, ml başına 1.0E + 07’den az P. jirovecii genomu olan birkaç PCR ürünü, PacBio dizilemesi için yeterli DNA elde etmek amacıyla E-gel adımından sonra bir araya getirilmeliydi.
PacBio dairesel konsensüs dizilemesi (CCS)
PCR ürünlerinin tek molekül gerçek zamanlı dizilenmesi, İsviçre’deki Lozan Üniversitesi Genomik Teknolojiler Tesisi’nde PacBio Sequel II kullanılarak gerçekleştirildi. Amplikonlara, birleştirilmeden önce barkodlar eklendi. Daha sonra, aynı msg -I geninin çoklu dizilemesini ve dairesel konsensüs dizilerinin oluşturulmasını sağlamak için daireselleştirildiler. Bu teknoloji, msg -I genlerinde bulunan tekrarlayan dizilerin uzun ve doğru okumalarını sağlar . 50 örneğin dizilenmesi (24 örnekten 48 PCR ürünü ve iki plazmid kontrolü) örnek başına 10.223 ila 160.836 okuma ile sonuçlandı.
PCR eserleri
Karışık alellerin PCR amplifikasyonu sırasında eserlerin üretimi ek biyolojik olmayan çeşitliliğe yol açar 55 . Koşullarımızda bu sorunun ortaya çıkmasını ele almak için, her biri tek bir spesifik msg -I aleli içeren iki plazmid PCR amplifikasyonundan önce veya sonra karıştırıldı ve daha sonra PacBio CCS kullanılarak dizilendi (çalışmada gözlemlenen tüm 1007 alel arasındaki %66’lık ortalamaya yakın olan %62 özdeşlik gösteren PL1c172754267 ve PL1u100008324 alellerini içeren PL1 ve PL2 plazmidleri [Ek Veri 5 ]). PCR’den önce karıştırıldığında kimerik amplikonlar gözlemlendi, ancak PCR’den sonra gözlemlenmedi. Okumaların çoğu plazmidlerde bulunan iki alele karşılık geldi (%92,5), %7,5’i muhtemelen bir PacBio hatasına karşılık gelen tek bir nükleotid polimorfizmi gösterdi ve %0,33’ü ilk veya son 500 bps’de tek bir rekombinasyonla açıklanabilen kimeralardı. Bu son PCR eserleri muhtemelen eksik iplikler üreten uzama adımı sırasında DNA polimerazının erken ayrılmasından kaynaklanır. Daha sonra bunlar yakından ilişkili dizilere bağlanarak primer olarak kullanılabilir. Eksik ipliklerin varsayılan uçları, kimerik dizilerin ilk ve son 500 bps’sinde lokalize edildi. Bu bulgular, yaklaşık 3,1 kb’lik dizilerin her iki ucunun kesilmesi ve sonraki analizler için yalnızca merkezi 2 kb’nin tutulması kararına yol açtı (pozisyonlar 500 ila 2500). Bu kırpma, bazı varyasyonları göz ardı ederek tanımlanan alellerin çeşitliliğini azalttı ancak yapay kimeralardan kaçınmak için gerekliydi. Sunulan sonuçları marjinal olarak etkiledi ve çıkarılan sonuçlar üzerinde hiçbir etkisi olmadı.
Alel tanımlama ve kantifikasyonu
Her bir numunede bulunan alelleri ve bunların bolluğunu belirlemek için Pacbio CCS ham okumalarının analizine yönelik özel bir biyoenformatik boru hattı geliştirildi. Yazılımın sürümleri ve paketleri hakkında bilgi Ek Tablo 2’de verilmiştir . Çok adımlı süreç aşağıdaki analiz dizisinden oluşmuştur:
- 1.hmmer 56, daha önce bir pilot deneyde tanımlanan ve CRJE’den sonra çerçeve içinde olmak için kırpılan 18 msg -I geni kullanılarak özel olarak oluşturulan bir profil-HMM’ye hizalayarak msg -I genlerine karşılık gelen okumaları tanımlamak için kullanıldı . nhmmer program komutu, okumaları hizalama uzunluğuna ve bit puanına göre filtrelemek için kullanıldı ve bu da okumaların yaklaşık %5’inin kaldırılması ve daha fazla analiz için örnek başına 9742 ila 156.375 okumanın son sayısının elde edilmesiyle sonuçlandı.
- 2.swarm (sürüm 3.1.0) 57 her örnek için küme tohumlarını tanımlamak için kullanıldı. Birbirinden 1 bp’den (seçenek -d 1) daha farklı olan her okuma swarm tarafından yeni bir küme tohumu olarak tanımlanır. Kümelerinde birden fazla okuma bulunan tohumlar, sonraki kümeleme adımı için küme tohumları olarak tutuldu.
- 3.cd-hit (sürüm 4.8.1) 58, swarm tarafından tanımlanan küme tohumları etrafında okumaları kümelemek için kullanıldı. -c 0.99 ve -g 1 seçeneklerine sahip cd-hit-est-2d komutu, benzer okumaların kümelerini oluşturmak ve bir sonraki bölümde açıklandığı gibi okumalar arasında tanımlanan alellerin bolluğunu belirlemek için her okumanın en benzer tohuma tahsis edilmesine izin verdi.
Şimdiye kadar elde edilen ince taneli kümeleme hala PacBio dizileme hatalarını kapsıyor ve bu sorundan kurtulmak için ek bir kümeleme adımı gerekiyordu. Her biri tek bir spesifik msg -I aleli içeren iki plazmit, biri UCS’li diğeri UCS’siz, PCR ile çoğaltıldı, PacBio CCS dizilendi ve yukarıda açıklandığı gibi kümelendi ve birkaç tohum dizisi elde edildi. Ancak, tek bir plazmitten gelen tohum dizileri arasındaki tüm çiftler halinde dizi kimlikleri %99,5’ten yüksekti ve bu da PacBio CCS dizilemesinin beklenen hata oranıyla uyuşuyordu 59 . Kimlik eşiği %99,5 olarak ayarlanarak ek bir kümeleme turu gerçekleştirildi. Bu, özdeş aleller veya kullanılan dizileme tekniği ile ayırt edilemeyen aleller olarak kabul edilen okuma kümeleri üretti. Bu, kırpılan 2 kb dizilerde 10 bps’den daha az bir farka karşılık gelir.
msg -I alel repertuarlarının doğrulanması
Gözlemlenen repertuarlar, beş hasta arasında verilen üç alele özgü PCR’ler kullanılarak desteklendi (Ek Tablo 3 , Ek Şekil 2 ). Birkaç hastada bulunan ve diğerlerinde bulunmayan üç alel seçildi. Her birini özgül olarak çoğaltmak için bu aleller içinde primerler tasarlandı (Ek Tablo 3 ). Primer çiftlerinin özgüllüğü, bunların mevcut çalışmada gözlemlenen genomik artı ifade edilen repertuarlarda tanımlanan 1007 farklı msg- I aleline ve tüm nükleotid koleksiyonuna (nr/nt) karşı patlatılmasıyla değerlendirildi. PCR’ler aşağıdaki parametrelerle gerçekleştirildi: 94 °C’de 3 dakika, ardından 94 °C’de 15 saniye, 56 °C’de 30 saniye ve 72 °C’de 2 dakikadan oluşan 35 döngü, ardından 72 °C’de 7 dakikalık son bir uzatma. PacBio CCS ile gözlenen hastalardaki varlığı veya yokluğu, spesifik PCR’ler ile doğrulandı (Ek Şekil 2 ). Sanger teknolojisi kullanılarak elde edilen amplikon dizileri, CCS kullanılarak elde edilenlerle %100 özdeşlik gösterdi. Pozitif DNA kontrolü, GenomiPhi HY kiti (GE Healthcare) kullanılarak hasta LA2’nin BAL’ından 2,5 µl DNA’nın rastgele amplifikasyonu ile üretildi, ardından QIAamp DNA Blood Mini Kit (Qiagen) kolonları kullanılarak bir saflaştırma adımı gerçekleştirildi. Ayrıca PCR reaksiyonlarının kurulumu için de kullanıldı.
P. jirovecii suşlarının sayısının tahmini
Netlik sağlamak adına, in vitro kültür olmadan tanımlanan her genotipin biyolojik bir suşa uygunluğunun belirlenememesi durumunda bile, bu yazıda “suş” terimini kullanıyoruz. Her bir hastayı enfekte eden P. jirovecii suşlarının sayısı, ribozomal RNA operonunun (ITS1-5.8S-ITS2) dahili transkripsiyonlu aralayıcıları ve 5.8S rRNA genini içeren bölgenin çoğaltılması ve dizilenmesiyle tahmin edildi. PCR karışımları, toplam 20 µl hacimde bir mikrolitre DNA ile msg -I genlerinin çoğaltılması için kullanılan karışımlarla aynıydı. PCR’ler aşağıdaki parametrelerle gerçekleştirildi: 95 °C’de 3 dakika ilk denatürasyon, ardından 95 °C’de 30 saniye, 62 °C’de 30 saniye ve 72 °C’de 45 saniyeden oluşan 35 döngü, ardından 72 °C’de 1 dakikalık son uzama. Primerler, 60 : 5′ GCTGGA AAGTTGATCAAATTTGGTC 3′ ve 5′ T TCGGACGAG AC T A CTCGCC 3′ referansında açıklanan primerlerden türetildi (altı çizili altı baz, tavlama sıcaklığını ayarlamak için ilk primere 5’te eklendi ve altı çizili dört baz, aslında ikinci primer içinde bulunan sıçanları enfekte eden tür olan P. carinii’den gelen bazların yerini aldı). PCR programı, 95 °C’de 3 dakikalık ilk denatürasyon, 95 °C’de 30 saniye, 62 °C’de 30 saniye ve 72 °C’de 45 saniyeden oluşan 30 ila 35 döngü ve ardından 72 °C’de 1 dakikalık son uzamayı içeriyordu. Qiagen PCR kolon kiti kullanılarak saflaştırıldıktan sonra, amplikonlar PacBio CCS kullanılarak dizilendi. ITS1-5.8S-ITS2 okumalarının analizine adanmış biyoenformatik boru hattı, doğru genleri tanımlamak için hmmer ile başladı ve ardından 470 ile 500 bps arasındaki okuma boyutuna göre bir filtreleme adımı izledi (yukarıya bakın, Alel tanımlama ve kantifikasyonu). Xue ve ark. 60 . ITS1-5.8S-ITS2 bölgesinin hem amplifikasyon hem de dizileme sırasında hataya eğilimli yedi homopolimer uzantısı içerdiğini vurguladı. Bu nedenle, verilerimizde değişiklik gösteren altı homopolimerin uzunluğu homojenleştirildi (Ek Şekil 3 ). Daha sonra yedekli okumalar swarm (seçenek d0) kullanılarak kümelendi ve yalnızca her hastada bulunan okumaların %1’inden fazlasını oluşturan kümeler analiz edildi. Gözlemlenen 25 genotipin dizilerinin GenBank’ta bulunan dizilerle ilişkisi Ek Tablo 4’te verilmiştir .
BLASTn analizleri
PacBio dizilemesi 8 kullanılarak tek bir suştan bir araya getirilen tüm P. jirovecii genomunda bulunan 37 subtelomer içindeki benzerlikleri aradık . NCBI web sitesindeki ( https://blast.ncbi.nlm.nih.gov/Blast.cgi ) BLASTn algoritmasını varsayılan parametre değerleri (beklenen eşik değeri 0,05) ve aşağıdaki ayarlarla kullandık : veritabanı: tüm genom shotgun kontigleri (wgs), sınırla: BioProject, BioProject adı: 382815 Pneumocystis jirovecii suşu E2178, program seçimi: biraz benzer diziler (BLASTn). Msg genlerini taşıyan 37 kontig, P. jirovecii genomunda bulunan 34-40’ın çoğuna (17-20 kromozom) karşılık gelen tüm veya kısmi subtelomerlerdir 4 . Bununla birlikte, 37 kontigin 17’si subtelomeri çevreleyen msg olmayan genler taşımaz , bu nedenle daha önce tanımlanmış belirli bir kromozoma atfedilemezler 4 . Sonuç olarak, bunlardan bazıları farklı olanlar yerine aynı subtelomerin parçaları olabilir. Basitleştirme için, her bir kontig, mevcut yazıda belirli bir subtelomer olarak kabul edilir.
Msg -I alelleri içinde mozaikçiliğin aranması
Bu makalenin genomik repertuarlarında tanımlanan 917 alel içinde mozaikliği aramak için çoğaltılmış parçalar kullanıldı. Rastgele seçilen bir alel, 917 alelin 3,1 kb dizileri arasında iki veya daha fazla diziyi hizalamak için varsayılan parametrelerle BLASTn kullanılarak patlatıldı (tam boyutlu aleller kullanıldı çünkü çoğaltılmış parçaların aranması nihai kimera tarafından engellenmez). En yakın aleli, yani en yüksek puana sahip olan, sonraki adımlar için seçildi. Her iki dizinin hizalanması, iki alel tarafından paylaşılan 100 bps’den daha büyük parçaların görsel olarak tanımlanmasına izin verdi. Daha sonra bu parçalar BLASTn kullanılarak 917 alel arasında arandı ve tamamen korunmuş parçaları içerenler belirlendi.
Biyoenformatik, bölgeye özgü rekombinasyonları araştırıyor
P. jirovecii genomunda (erişim numarası LFWA01000000 ) siteye özgü rekombinasyonları kodlayan potansiyel genler, bu genomu tBLASTn (NCBI BLAST paketi) kullanarak temsili yem dizilerinin büyük havuzlarıyla eşleştirerek arandı. Bu havuzlar, geniş bir takson yelpazesinden InterPro açıklamaları IPR011010 (DNA parçalama-yeniden birleştirme enzimi, katalitik çekirdek) aracılığıyla işe alındı. Kullanılan yemlerin UniProt tanımlayıcıları P03870, P13769, P13770, P13783, P13784 ve P13785 idi. Çok katı koşulların kullanımı nedeniyle adayların kaybolmasını önlemek için, tBLASTn aramaları gevşetilmiş parametrelerle (1E-4’ten varsayılan değere kadar E değeri) gerçekleştirildi. Uygun bir E değerine sahip her eşleşme, önceden var olan açıklamaları arayarak araştırıldı. Kodlama dizisi (CDS) açıklamaları mevcut değilse, eşleşen bölge varsayımsal yeni CDS’ler açısından değerlendirildi ve çevrilen dizileri gerekli referans imza eşleşmelerini tespit etmek için InterProScan4 aracına gönderildi 26 .
Kaynak ve devamına Buradan ulaşabilirsiniz.