Ekzokrin ve endokrin bozukluklar ve yetersizlikler, kronik pankreatitte (CP) iki önemli zararlı patolojik süreçtir ve steatore ve diyabete yol açabilir. Bununla birlikte, CP’de ekzokrin ve endokrin bozuklukların şiddetini değerlendirmek için güvenilir klinik sınıflandırma şemaları eksiktir ve altta yatan mekanizmalar da belirsizdir. Özellikle, CP’de ekzosom bazlı sıvı biyopsi ve sınıflandırma eksiktir. Bu çalışmada, farklı CP şiddet derecelerine sahip CP hastalarından alınan plazma eksozomlarında transkriptom dizilemesi gerçekleştirdik. Ayrıca, sınıflandırmayı yorumlamak için CP hastalarındaki pankreas lezyonlarından alınan tek hücreli dizileme verilerini analiz ettik ve sınıflandırmayı doğrulamak için harici bir kohort oluşturuldu.
Sonuç olarak, plazma eksozomlarındaki 12 mikroRNA ekspresyonuna dayanarak CP hastalarında steatore ve diyabet başlangıcını tahmin etmek için 3 aşamalı bir sınıflandırma sistemi oluşturduk ve ön doğrulamasını yaptık. Bu sınıflandırma sistemini uygulayan halka açık bir çevrimiçi araç da geliştirildi. CP farelerinden alınan tek hücre dizileme verileriyle birlikte yapılan ileri analizler, ekzozom kaynaklı miR-24-3p ve nötrofil S100A8’in CP ilerlemesinde önemli faktörler olduğunu belirlemiştir. Mekanik olarak, bulgularımız CP’de aşağı regüle edilen ekzozom kaynaklı miR-24-3p’nin nötrofillerde hedef geni S100A8’in yukarı regülasyonuna yol açabileceğini ve böylece pankreas yıldız hücrelerinin fibrotik fenotipini aktive ederek ve makrofajlarda inflamasyona neden olarak pankreas β hücrelerinin apoptozuna yol açarak CP ile ilişkili ekzokrin ve endokrin bozuklukları teşvik edebileceğini göstermektedir. Çalışmamız birlikte, CP için yeni bir ekzozom tabanlı 3 aşamalı sınıflandırma sistemi sunmakta ve ekzozomal miR-24-3p ve S100A8’in fibrozis ve pankreas β hücresi apoptozundaki rolünü vurgulamaktadır.
giriiş
Kronik pankreatit (KP), pankreas dokusunu etkileyen ve genetik ve çevresel faktörlerin karmaşık bir etkileşimi tarafından yönlendirilen, giderek kötüleşen fibroz ve inflamasyon durumudur 1 . KP, pankreas parenkiminin fibröz doku ile yer değiştirmesine yol açan tekrarlayan inflamasyon ataklarıyla karakterizedir. Bu fibroz, hem ekzokrin hem de endokrin pankreas fonksiyonlarında kademeli bir düşüşe neden olarak psödokist oluşumu, pankreas ve safra kanallarının tıkanması, steatore, diyabet, vasküler sorunlar, yetersiz beslenme ve sürekli ağrı dahil olmak üzere çeşitli komplikasyonlara yol açar. Bu komplikasyonlar arasında steatore ve diyabet özellikle ciddidir. Steatore, ciddi pankreas ekzokrin yetmezliğinin önemli bir belirtisidir ve genellikle besin emiliminde malabsorpsiyonla birlikte görülür, bu da vitamin ve mikro besin eksikliğine ve kilo kaybına yol açar ve erken ateroskleroz, kardiyovasküler olaylar, osteoporoz, kırık, bağışıklık yetersizliği ve enfeksiyon geliştirme riskini artırır 2 , 3 , 4 , 5 . CP’nin gelişmesinden sonra ortaya çıkan diyabet mellitusa tip 3c diyabet mellitusu (T3cDM) denir ve endokrin disfonksiyonunun bir işaretidir 2. Tüm diyabet hastalarını etkileyen ciddi komplikasyonların yanı sıra, T3cDM’li CP hastaları hipoglisemi olaylarına bağlı komplikasyonlara veya ölüme karşı özellikle savunmasızdır 6 , 7. Bu nedenle CP, etkilenen hastaların hem yaşam kalitesini hem de yaşam beklentisini önemli ölçüde azaltır.
SP’nin etki yelpazesi göz önüne alındığında, hastalığın şiddetini değerlendirmek, tedavi etkilerini izlemek ve ciddi komplikasyon riskini belirlemek bu hastalıkta özel bir öneme sahiptir. 1963’te Marsilya’da bir sempozyumda oluşturulan ilk SP sınıflandırması, morfolojik değişiklikleri ve hastalık etyolojisini vurgulamıştır 8 . 1985’te revize edilen bir versiyon, bu morfolojik kriterleri iyileştirmiş ve bunları pankreas fonksiyonundaki olası kayıplarla ilişkilendirmiş, Marsilya-Roma sınıflandırmasına daha fazla alt tip eklemiştir 9 , 10 . CP için ilk klinik derecelendirme sistemi olan Cambridge sınıflandırması, endoskopik retrograd pankreatografide (ERP) ve bir dereceye kadar ultrason veya BT bulgularında gözlenen kanal değişikliklerine dayanmaktadır 11 , 12 . Cambridge kriterleri, SP’nin görüntüleme tabanlı derecelendirmesinde standart olmasına rağmen, uzmanlar bu morfolojik değişikliklerin pankreasın işlevsel veya histolojik durumunu doğru bir şekilde yansıtmayabileceğini ve potansiyel olarak erken hastalık evrelerini gözden kaçırabileceğini belirtmişlerdir 13 . Daha yakın zamanlarda, hastalığın ilerlemesini görüntüleme, klinik semptomlar, laboratuvar indeksleri, müdahale ihtiyacı ve işlevsel kayıp temelinde derecelendirerek tanımlamak için çeşitli sınıflandırma sistemleri geliştirilmiştir 14 , 15 , 16 , 17 , 18. Ancak, bu sistemler CP şiddetinin derecesini moleküler biyoloji özelliklerinin bir kombinasyonu yerine yalnızca klinik bir indeks temelinde değerlendirmek üzere tasarlanmıştır ve bu da bu sınıflandırmaların klinik doğruluğunu tehlikeye atar.
Son 20 yılda elde edilen moleküler içgörüler CP’nin heterojen doğasını göstermiştir. Özellikle, birkaç genetik mutasyonun tripsin düzenlenmesini bozarak, anti-proteolitik savunmaları zayıflatarak veya kalsiyum sinyallemesini değiştirerek hastalık patogenezine katkıda bulunduğu bulunmuştur. Örneğin, PRSS1 işlev kazanımı mutasyonları kalıtsal CP’nin ayırt edici itici güçleridir 19 , buna karşın CASR polimorfizmleri alkolle ilişkili alt tiplerle güçlü bir şekilde ilişkilidir 20 . Bu bulgulara dayanarak, son çalışmalar CP’yi genetik, alkolle ilişkili ve idiyopatik alt tiplere ayırmak için moleküler alt tipleme stratejileri önermiştir 1 . Bununla birlikte, bu sınıflandırma sistemleri büyük ölçüde etiyoloji tabanlı kalırken, çoklu-omik biyobelirteçlerin sistematik olarak entegre edilmesi, özellikle steatore ve diyabet olmak üzere mevcut komplikasyon riskinin hassas bir şekilde tabakalandırılmasına ve değerlendirilmesine olanak sağlayacaktır. Ayrıca, yeni belirteçlere ve yüksek verimli dizilemeye dayalı sıvı biyopsi teknikleri bu sistemlerde uygulanmamıştır. Ayrıca, hastalığın şiddetinin farklı düzeyleriyle ilişkili temel mekanizmalar iyi tanımlanmamıştır.
Hücre dışı veziküller (eksozomlar), lipit çift tabakası ve tipik boyutları 50 nm ila 1 µm olan parçacıklardır; kargo olarak çoğunlukla proteinler ve mRNA’lar, uzun kodlamayan RNA’lar (lncRNA’lar) ve miRNA’lar dahil RNA’lar içerirler. Daha önceki birkaç çalışma, pankreas parenkimal hücreleri tarafından üretilen eksozomların pankreas hücresi iletişiminin temel aracıları olduğunu 21 ve CP’de fibrozis 22 , 23 ve ekzokrin ve endokrin bozukluklarda 24 rol oynadığını göstermiştir . Bununla birlikte, periferik eksozomların klinik açıdan önemli RNA ve protein biyobelirteç kaynakları olarak potansiyeli büyük ölçüde araştırılmamıştır.
Bu çalışmada, CP’deki hasarlı pankreas parenkimal hücreleri tarafından salınan spesifik protein ve RNA’ların dolaşımdaki ekzosomlar tarafından kapsüllendiğini ve bu hücrelerde tanımlanabileceğini ön olarak doğruladık. Bu ekzosomlar, CP’deki çeşitli hasarlı pankreas parenkimal hücre tiplerinin transkriptomik ve proteomik karakterizasyonuna olanak tanır. Bu yaklaşım, hastalığın şiddetini ve komplikasyon riskini değerlendirmek ve CP ile ilişkili ekzokrin ve endokrin işlev bozukluğunun altında yatan mekanizmaları aydınlatmak için yapay zekâ tabanlı bir tabakalandırma sisteminin geliştirilmesi için potansiyel göstermektedir.
Sonuçlar
Tüm transkriptom dizilemesi, CP hastalarında ve sağlıklı donörlerde farklı şekilde ifade edilen ekzosom kaynaklı RNA’ları ortaya koyuyor
Çalışma tasarımının akış şeması Şekil 1a’da gösterilmiştir . İlk olarak, 89 CP hastasından ve 22 sağlıklı donörden (NC) plazma toplandı ve ekzosomlar çıkarılıp doğrulandı (Şekil 1b ; Ek Tablo S1 ve Şekil S1a, b ). Daha sonra, ekzosomların RNA içerikleri dizilendi ve hasta ve sağlıklı gruplar arasında mRNA’lar, miRNA’lar, lncRNA’lar ve dairesel RNA’ların (circRNA’lar) farklı ifade analizi yapıldı ve bunu yol zenginleştirme analizi izledi. Tüm RNA tipleri, CP hastaları ve NC’ler arasında farklı dağılımlar gösterdi (Şekil 1c, f, h , j). Farklı ifade edilen genler (DEG’ler), |logFold Change (FC) > 1| kriterlerine göre tanımlandı. ve yanlış keşif oranı (FDR) < 0,05 ve görselleştirildi (Şekil 1d, k ; Ek Şekiller S1c , e, f, S2a, b, f ve Tablolar S2 – S5 ). DEG’ler arasında zenginleştirilen yollar, çeviriyle ilgili yolların ve keratinosit göçünün teşvik edildiğini göstermektedir (Şekil 1e ; Ek Şekil S1d ). Farklı şekilde ifade edilen lncRNA’ların (DElncRNA’lar) varsayımsal cis – ve trans -düzenleyici mRNA’ları arasında zenginleştirilen yollar otizm ve nötrofil aktivasyonu ile ilişkiliydi (Şekil 1g ) ve Gen Ontolojisi (GO) analizi DNA replikasyon yollarının aktivasyonunu önerdi (Ek Şekil S1g, h ). Bu yollardaki DElncRNA’ların varsayımsal cis – ve trans -düzenleyici mRNA’ları da görselleştirildi (Ek Şekil S1i–k ).

Daha sonra, farklı şekilde ifade edilen circRNA’ların konak genleri tanımlandı. Zenginleştirilmiş GO terimleri endositozla ilişkiliydi (Şekil 1i ; Ek Şekil S2d, e ). Zenginleştirilmiş yol terimleri protein süreçleriyle ilişkiliydi (Ek Şekil S2c ). Ek olarak, yol zenginleştirme analizi, farklı şekilde ifade edilen miRNA hedefli genlerin FoxO, MAPK, Erbb ve Hippo yolları, endokrin direnci ve insülin sinyal yolu dahil olmak üzere çeşitli spesifik yolların aktivasyonuyla ilişkili olduğunu ortaya koydu (Şekil 1l ; Ek Şekil S2h–k ). GO analizi ayrıca protein süreciyle ilgili terimlerin zenginleştirildiğini de ortaya koydu (Şekil 1g ).
Üç seviyeli bir sınıflandırma komplikasyon tahmin sisteminin kurulması ve doğrulanması
Önceki bir çalışma, lezyondan türetilen eksozomların periferik kana yayılabileceğini ve bu eksozomları ölçmek için sıvı biyopsinin prostat kanseri hastalarında değerli olduğunu göstermiştir 25 . Bu nedenle, CP’deki yaralı pankreas parenkimal hücrelerinden gelen RNA ve proteinlerin eksozomlar içinde kapsüllenmiş ve daha sonra kan dolaşımına salgılanmış olabileceğini tahmin ediyoruz. Özellikle, periferik eksozomların RNA içeriğinin CP hastalarında pankreas parenkimal hücre hasarının şiddetini ve steatore ve diyabet dahil CP ile ilişkili komplikasyon riskini yansıtabileceği hipotezini kurduk. Hipotezimizi test etmek için, öncelikle herkese açık bir veritabanından indirilen tek hücreli dizileme verilerini yeniden analiz ettik. Çevrimiçi tek hücreli dizileme verilerinin şeması Şekil 2a’da gösterilmektedir 26 . Açıklama, CP grubunda 13 hücre tipini tanımladı (Şekil 2b ; Ek Şekil S3a–c ) ve ilgili işaretleyici genlerin ifade düzeyleri görselleştirildi (Şekil 2c ). Daha sonra, tek hücre dizileme verilerinde iki gen imzasının ifadesini tanımladık ve görselleştirdik: asiner REG + hücrelerinin ve aktive yıldız hücrelerinin en iyi 40 işaretleyici geninden (logFC değerlerine göre) oluşan bir ekzokrin imza (Ek Tablo S6 ) ve alfa hücrelerinin ve beta hücrelerinin en iyi 40 işaretleyici geninden (logFC değerlerine göre) oluşan bir endokrin imza (Şekil 2d ; Ek Tablo S7 ). İki imza, ekzosom dizileme verilerimize eşlendi ve daha sonra CP hastalarında her imzadaki genlerin medyan ifade düzeyleri, her imzanın ifadesi olarak tanımlandı ve nokta grafiğinde görselleştirildi (Şekil 2e ). Özellikle, hem ekzokrin imza genleri hem de endokrin imza genleri sağlıklı donörlerde düşük seviyelerde, CP hastalarında ise daha yüksek seviyelerde ifade edildi; bu, ilk varsayımlarımızla tutarlıydı.

Sonra, CP hastalarının periferik kanındaki ekzosomlardaki farklı RNA tiplerinin kombinasyonlarına dayalı ortak alt tipleri tanımlamak için, yukarıdaki dört moleküler tipin her birine (mRNA, miRNA, lncRNA ve circRNA) küme ataması (COCA) algoritması 27 ile analiz uygulandı. Küme numarası, en büyük silüet puanını ve en düşük PAC puanını ürettiği için 3 olarak ayarlandı (Ek Şekil S3d ). 3 küme, COCA1–3, CP hastası alt tipleri olarak tanımlandı (Şekil 2f ). Her bir CP hastası kümesinin klinik özellikleri Ek Tablo S8’de gösterilmiş olup , COCA1’den COCA3’e doğru diyabet mellitus ve steatore morbiditesinin artmasıyla birlikte CP şiddetinin artma eğilimini göstermektedir. Bu bulguyla tutarlı olarak, COCA alt tiplerinin dönüşümünün ekzokrin ve endokrin imzaların artan ifadesiyle aynı zamana denk geldiğini de gözlemledik (Şekil 2g, h ). Ekzosom dizileme verilerindeki bu iki imzadaki genlerin ifadesinin PCA’sı ayrıca COCA1 kümesinin normal örneklere (sağlıklı donörler) daha yakın olduğunu, COCA2 ve COCA3 kümelerinin ise normal örneklerden daha uzakta olduğunu ortaya koydu (Ek Şekil S3e ).
Yukarıdaki bulgulara dayanarak, bu COCA sınıflandırma sisteminin SP hastalarında diyabet ve steatore riskini tahmin etmek için kullanılıp kullanılamayacağını belirlemeyi amaçladık. Bu amaçla, örnek toplama sırasında steatore veya diyabeti olmayan hastalar takibe dahil edildi. Bu hastalar arasında, COCA3 veya COCA2 alt tiplerine sahip olanlarda, COCA1 alt tipine sahip olanlara kıyasla anlamlı derecede daha yüksek steatore ve diyabet insidansı gözlemledik (Şekil 2i, j , P < 0,05). Bu da geliştirdiğimiz COCA sisteminin SP hastalarında başlangıç aşamasındaki diyabet ve steatore riskini tahmin edebileceğini düşündürmektedir.
Her COCA alt tipinin ekzosomla ilişkili temel özelliklerinin tanımlanması
Ekzosom dizilemesinden elde edilen mRNA verilerine dayanarak her COCA alt tipinde aktive olan temel yolları tanımlamak için ağırlıklı gen korelasyon ağı analizi (WGCNA) gerçekleştirdik (Ek Şekil S4a–d ). On sekiz ayrı modül tanımlandı (Şekil 3a ) ve korelasyon analizi MElightyellow, MEturquoise ve MEroyalblue modüllerinin sırasıyla COCA1, COCA2 ve COCA3 alt tipleriyle anlamlı derecede pozitif korelasyonlu olduğunu ortaya koydu (Şekil 3b ). Dolayısıyla, bu modüller her bir ilgili COCA alt tipine özgü belirteç modülleri olarak tanımlandı. COCA1 alt tipinde zenginleştirilen yolaklar (MElightyellow modülü), savunma tepkisi ve hücre dışı uyarı dahil olmak üzere stres tepkileriyle ilişkiliydi (Şekil 3c ). COCA2 alt tipinde zenginleştirilen yolaklar (MEturquoise modülü), peptit metabolik süreçleri ve mRNA metabolik süreçleri dahil olmak üzere metabolik değişikliklerle ilişkiliydi (Şekil 3d ). COCA3 alt tipinde (MEroyalblue modülü) zenginleştirilmiş yollar, nötrofille ilişkili göç ve kronik inflamatuar yanıt dahil olmak üzere inflamasyonla ilişkiliydi (Şekil 3e ). MEroyalblue’daki en çok yukarı regüle edilen 17 gen Şekil 3f’de gösterilmiş olup , normal gruptan COCA1 grubuna ve ardından COCA3 grubuna doğru giderek artan bir eğilim göstermektedir. Daha sonra, deneysel kanıtlara dayalı olarak bu gruplar içindeki aday miRNA-mRNA etkileşim ağları miRTarBase’den indirilmiştir (Ek Tablo S9 ). Bu genlerin potansiyel olarak süngerlenmiş önemli ölçüde aşağı regüle edilmiş miRNA’ları Şekil 3g’de gösterilmiştir .
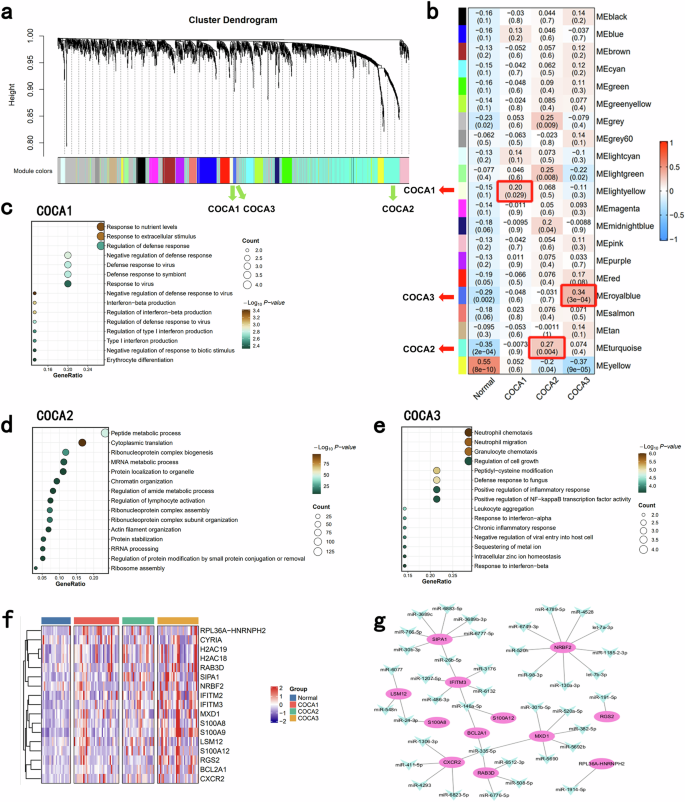
12-miRNA tanı sınıflandırıcısı kontrol, COCA1, COCA2 ve COCA3 hastalarını doğru bir şekilde ayırır
Yukarıdaki yaklaşım ekzosom dizileme verilerine dayanarak CP hastalarının üç alt tipini tanımlasa da, hala birkaç metodolojik eksiklik vardı. Özellikle, bu sınıflandırma algoritması sağlıklı bireyleri CP hastalarından ayırt edemedi ve hassas sınıflandırma için yalnızca birkaç belirteç yerine kapsamlı ekzosom dizileme verileri gerektirdi. Ek olarak, alt tipler arasındaki klinik farklılıklar doğrulama kohortunda değerlendirilmedi. miRNA’ların ekzosomlarda nispeten kararlı olduğunu ve optimum tanı indeksleri olduğunu bildiren önceki çalışmalar ışığında 28 , daha sağlam bir tanı geliştirmek için bu RNA’lara odaklandık. İlk olarak, ekzosom miRNA dizileme verilerinde bulunan yüksek boyutluluk ve çoklu doğrusallık zorluklarını ele almak için, seyrek, ilişkili biyolojik veri kümelerinde biyobelirteç seçimi için etkili olduğu kanıtlanmış bir yöntem olan L1 düzenlemesiyle LASSO regresyonunu uyguladık 29 . LASSO regresyondan türetilen biyobelirteçler dikkat çekici klinik çevrilebilirliğe sahiptir ve BPNN’nin doğrusal olmayan modelleme yetenekleriyle entegre edildiğinde hassas hastalık alt tipi tabakalaşmasına olanak sağlayabilir; bu sinerjik yaklaşım yalnızca hesaplamalı karmaşıklığı azaltmakla kalmaz, aynı zamanda tanı çerçevesinin biyolojik yorumlanabilirliğini de artırır. Bu nedenle, mevcut COCA alt tiplerinden ve normal örneklerden elde edilen miRNA dizileme verilerine dayanarak LASSO + BPNN algoritmasını kullanarak 12-miRNA tanı sınıflandırıcısı oluşturduk (Şekil 4a ). 12 miRNA’nın ifadesi Şekil 4b’de gösterilmiştir ve BPNN’nin yapısı Şekil 4c’de görselleştirilmiştir . Dizileme verileri daha sonra rastgele bir eğitim setine (%80 hasta) ve bir test setine (%20 hasta) ayrıldı. Sınıflandırıcının etkinliği daha sonra eğitim setleri, test setleri ve bunların kombinasyonları (tüm hastalar) üzerinde test edildi ve %85-96,7 doğruluk değerleri elde edildi (Şekil 4d ). Daha sonra, bu 12 miRNA’nın kantitatif polimeraz zincir reaksiyonu (qPCR) için tüm ekzosom dizileme örneklerinden rastgele 53 örnek seçildi (Şekil 4e ); qPCR sonuçları daha sonra sınıflandırıcıya girildi ve %83 doğruluk elde edildi (Şekil 4f ). Daha sonra, sağlıklı kontrollerden ( n = 10) ve başka bir kohorttan CP hastalarından ( n = 90) alınan plazma ekzosom örneklerinde PCR gerçekleştirdik ve bu örnekler arasındaki COCA alt tiplerini tahmin ettik (Ek Tablolar S10 , S11 ). Sağlıklı bireylerden ( n = 10) alınan tüm normal örnekler doğru bir şekilde tanımlandı.
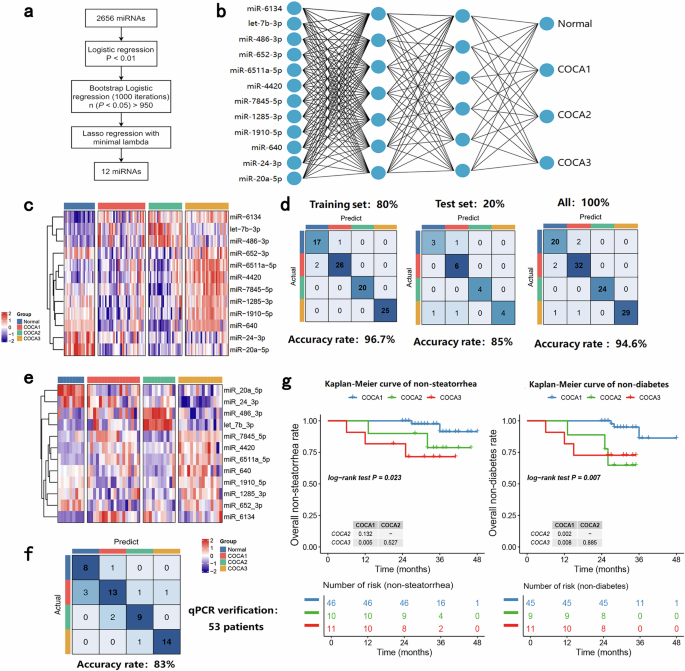
Her kümenin klinik özelliklerinin analizleri ayrıca COCA1’den COCA3’e doğru diyabet ve steatore morbiditesinde bir artış olduğunu gösterdi (Ek Tablo S12 ). Dahası, örnek toplama sırasında steatore veya diyabeti olmayan hastaların takibi, COCA3 veya COCA2 alt tipine sahip hastalarda steatore ve diyabet insidansında COCA1 alt tipine sahip olanlara kıyasla önemli bir artış olduğunu ortaya koydu (Şekil 4g , P < 0,05). CP için sınıflandırma sisteminin tanısal faydasını daha da doğrulamak için, 15 osteoartrit vakası ve 3 romatoid artrit vakasını temsil eden halka açık veritabanlarından (GSE263996, GSE218599) miRNA verilerini indirdik. Toplu etki kaldırıldıktan sonra, bu veri kümeleri 89 CP hastasından oluşan yerel keşif kohortumuzun miRNA verileriyle birleştirildi. Daha sonra COCA sınıflandırıcısı, 12 önceden tanımlanmış miRNA’nın ifade düzeylerine dayanarak CP durumunu tahmin etmek için uygulandı.
107 denek (89’u CP’li ve 18’i diğer hastalıklara sahip) arasında, öngörülen 89 CP vakasından 78’i COCA algoritması tarafından doğru bir şekilde tespit edildi (duyarlılık: %87,6, özgüllük: %77,8; Ek Tablo S13 ). Bu sonuçlar, algoritmamızın yalnızca CP ile ilişkili komplikasyonlar (örneğin, steatore ve diyabet) için öngörücü etkinlik göstermekle kalmayıp, aynı zamanda CP’yi CP dışı durumlardan ayırt etmede potansiyel tanı değerine sahip olduğunu önsel olarak göstermektedir.
CP tanı sınıflandırıcısında ekzosom kaynaklı miR-24-3p ile S100A8 arasındaki etkileşimin temel rolünün belirlenmesi ve CP’de ekzosomal S100A8 kaynağı olarak nötrofillerin kullanılması
miRNA’lar ve hedef genlerinin ekzosom kaynaklı hastalık ilerlemesinin temel mekanizmaları olarak bildirildiği göz önüne alındığında 28 , CP ilerlemesinde 12 miRNA’nın mekanizmasını daha fazla açıklamaya çalıştık. Tanı panelimizdeki 12 miRNA’nın potansiyel etkileşim ortağı olarak tanımlanan 2750 mRNA’yı çıkardık (Ek Tablo S9 ); bu 2750 gen Ek Tablo S14 ‘ te listelenmiştir . Daha sonra, Şekil 5a ‘da gösterildiği gibi, bu 12-miRNA COCA tanı sınıflandırıcıdaki tahmin edilen miRNA-mRNA çiftlerini miRNA-mRNA etkileşim tahmini ve korelasyon analizi yoluyla filtreledik. Özellikle, miR-24-3p, miR-20a-5p ve bunların varsayımsal mRNA ağları , temel faktörler olarak tanımlandı (Şekil 5b ; Ek Şekil S5a solda ve Tablo S15 ). miR-24-3p ifadesinin normal örneklerden COCA1–3 örneklerine doğru miR-20a-5p’ye kıyasla daha tutarlı bir düşüş eğilimi göstermesi nedeniyle (Şekil 5c ; Ek Şekil S5a sağda), sonraki deneyler için miR-24-3p seçildi. Benzer şekilde, miR-24-3p’nin varsayılan hedef mRNA’ları arasında, yalnızca S100A8 normal örnekler ve COCA1–3 örnekleri arasında tutarlı bir yükseliş eğilimi gösterdi (Şekil 5d ; Ek Şekil S5b ). Bu nedenle, daha sonra miR-24-3p ve S100A8 arasındaki etkileşimin CP gelişimini etkileyip etkilemediğine odaklandık.
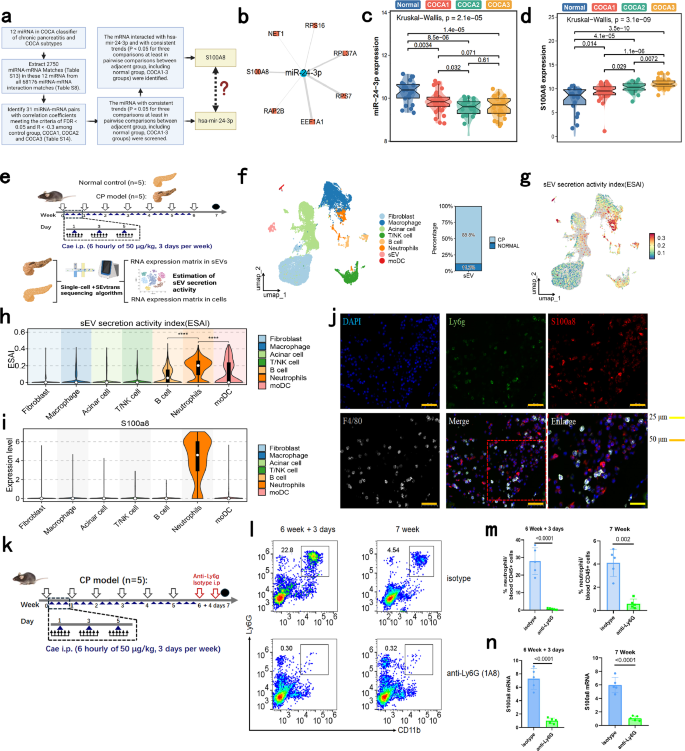
İlk sorumuz, CP’de hangi hücre tipinin öncelikli olarak eksozomları serbest bıraktığıydı. Yukarıda gösterildiği gibi, CP’de hasarlı pankreas parenkimal hücreleri tarafından serbest bırakılan spesifik proteinlerin ve RNA’ların dolaşımdaki eksozomlarda tanımlanabileceğini ön olarak doğruladık. Bu nedenle, CP hastalarından ve normal kontrollerden alınan pankreas dokusunda tek hücre dizilemesi gerçekleştirdik. sEVtrans algoritması ayrıca tüm hücre tipleri arasında eksozom salgılama aktivite indeksini (ESAI) tahmin etmek için kullanıldı (Şekil 5e ). Ayrıca fare pankreas dokularından eksozomlar çıkardık ve bu örneklerde miR-24-3p ve S100A8 mRNA ifade seviyelerini ölçtük. İlk olarak, homolojilerini değerlendirmek için mir-24-3p dizileri fareler ve insanlar arasında karşılaştırıldı; bu karşılaştırma mmu-mir-24-3p dizisinin hsa-mir-24-3p dizisiyle aynı olduğunu gösterdi (Ek Şekil S5c ). Ekzosomlardaki mmu-mir-24-3p ekspresyonu CP grubunda normal (NC) gruba göre anlamlı derecede düşüktü ve CP ekzosomlarındaki S100A8 ekspresyonu CP grubunda anlamlı derecede daha fazlaydı (Ek Şekil S5d ).
Daha sonra, sEVtrans kalite kontrolü (Ek Şekil S5e, f ) ve hücre kümelemesi (Ek Şekil S5g, h ) kullanılarak ekzosom verilerinin ayrılmasının ardından tek hücre dizileme verilerine dayanarak hücre açıklaması gerçekleştirildi . Makrofajlar, T hücreleri, NK hücreleri, asiner hücreler, fibroblastlar, nötrofiller, monosit benzeri dendritik hücreler (moDC’ler), naif B hücreleri, plazma ve kırmızı kan hücreleri tanımlandı (Ek Şekil S5i ). Daha sonra sEVtrans’lardan elde edilen ekzosom verileri tek hücre dizileme verileriyle birleştirildi ve ekzosomların %88,8’inin CP grubundan türetildiği belirlendi (Şekil 5f ). Daha sonra, hücre tiplerinin ESAI değerleri sEVtrans kullanılarak belirlendi. Nötrofiller, çekirdek ekzosom salgılayan hücre tipi olarak tanımlandı (Şekil 5g , h). Ayrıca, S100A8 mRNA ekspresyonu yalnızca nötrofillerde tespit edildi (Şekil 5i ) ve S100A8 protein ekspresyonu Ly6g + nötrofillerde tespit edildi ancak F4/80 + makrofajlarda tespit edilmedi (Şekil 5j ).
Nötrofillerin ekzosomal S100A8 mRNA’sının kaynağı olduğunu doğrulamak için, CP’li bir fare modelinde nötrofil tükenmesi gerçekleştirdik (Şekil 5k ; Ek Şekil S5j ). 6 ve 6 hafta + 4 günde anti-Ly6g antikorunun intraperitoneal enjeksiyonu nötrofilleri başarıyla tüketti (Şekil 5l, m ). Daha sonra ekzosomları plazmadan çıkardık ve S100A8 ve mmu-mir-24-3p’nin ifade seviyelerini tespit ettik. S100A8 mRNA ifadesi, CP model farelerinde nötrofil tükenmesinden sonra önemli ölçüde azaldı (Şekil 5n ) ve mmu-mir-24-3p ifadesinde önemli bir değişiklik gözlenmedi (Ek Şekil S5k ). Dahası, CP model farelerde nötrofil tükenmesinden sonra patolojik skor önemli ölçüde azaldı (Ek Şekil S5l ). Bu sonuçlar bir araya geldiğinde, nötrofillerin CP’nin plazma ekzosomlarındaki S100A8 mRNA’sının kaynağı olmasına rağmen, ekzosomal miR-24-3p’nin nötrofiller tarafından üretilmediğini göstermektedir.
Ekzosom kaynaklı miR-24-3p kaybı, nötrofillerde S100A8 yukarı regülasyonuna yol açarak, pankreas yıldız hücrelerinde S100A8-TLR4/RAGE-ROS ekseni tarafından fibrotik fenotipi güçlendirir
Mir-24-3p ve S100A8’i CP hastalarının plazma eksozomlarında önemli bir etkileşim çifti olarak tanımladıktan ve eksozomal S100A8 mRNA’sının esas olarak nötrofillerden türetildiğini gösterdikten sonra, nötrofillerin mir-24-3p ve S100A8 etkileşiminin efektör hücreleri olduğu hipotezini ortaya koyduk. Bu nedenle, daha sonra nötrofillerin CP hastalarının plazmasından eksozomları alıp alamadığını test ettik. HL-60 hücrelerinde in vitro nötrofil benzeri bir fenotip başarıyla oluşturduk (Ek Şekil S6a ). Bu hücreler tarafından eksozom alımının daha sonra tespiti, CP hastalarının plazmasından türetilen eksozomların bu nötrofil benzeri HL-60 hücreleri tarafından alınabileceğini ortaya koydu (Şekil 6a ; Ek Şekil S6b ). Üstelik, bu hücrelerde miR-24-3p’nin yukarı düzenlenmesi, mRNA ve protein düzeylerinde S100A8 ekspresyonunu inhibe etti ve miR-24-3p baskılanması üzerine zıt etki gözlendi (Şekil 6b, c ; Ek Şekil S6c ). Ayrıca, periferik nötrofilleri CP hastalarından ve sağlıklı donörlerden başarıyla ayırdık (Ek Şekil S6d ). Eksozom alım tespiti ayrıca, CP hastalarının plazmasından türetilen eksozomların ayrılan nötrofiller tarafından alınabileceğini de ortaya koydu (Ek Şekil S6e ). Dahası, CP hastalarından alınan nötrofillerdeki S100A8 mRNA ve protein düzeylerinin sağlıklı kontrollerden alınan nötrofillerden önemli ölçüde daha yüksek olduğunu gözlemledik (Ek Şekil S6f, g ).
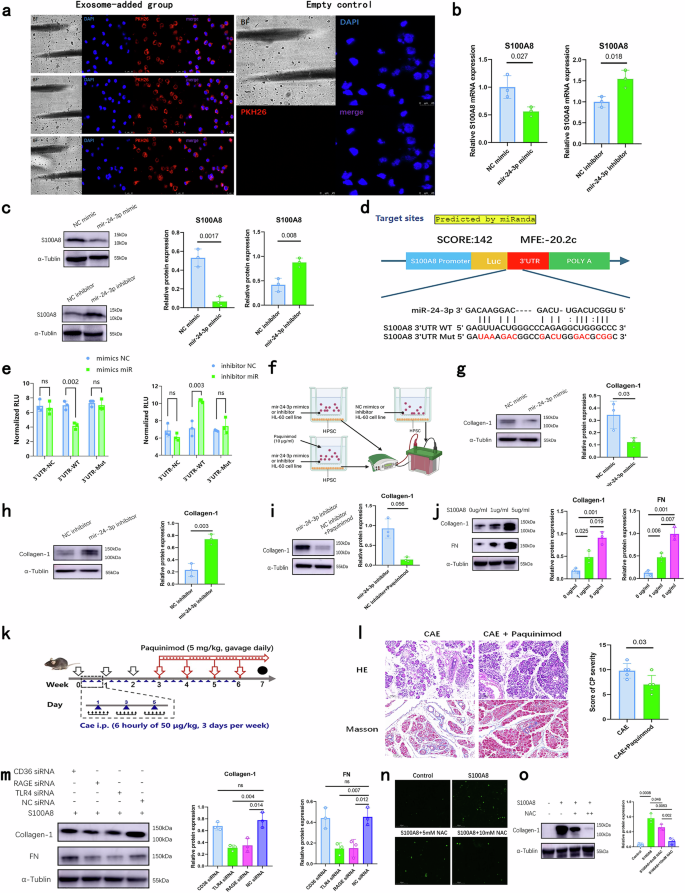
Daha sonra, miRanda yazılımı aracılığıyla miR-24-3p ve S100A8 arasındaki 3′-çevrilmemiş bölge (UTR) bağlanma bölgesini tahmin ettik ve S100A8’in 3′-UTR’sinde mutasyona uğramış ve vahşi tip (WT) bağlanma bölgeleri taşıyan lüsiferaz haberci vektörleri oluşturduk (Şekil 6d ). miR-24-3p’nin doğrudan S100A8’i hedef alıp alamayacağını daha ileri düzeyde test etmek için, bu lüsiferaz haberci vektörlerini HEK293T hücrelerine transfekte ettik. Lüsiferaz haberci deneyleri, WT 3′-UTR’nin lüsiferaz haberci aktivitesinin miR-24-3p taklitleri tarafından güçlü bir şekilde inhibe edildiğini ve miR-24-3p inhibitörü tarafından arttırıldığını ortaya koydu (Şekil 6e ).
Daha sonra, nötrofillerin insan pankreas yıldız hücreleri (HPSC’ler) üzerindeki profibrotik etkisini değerlendirmek için bir ortak kültür sistemi tasarlandı (Şekil 6f ). miR-24-3p’nin yukarı düzenlenmesi, kolajen-1 ekspresyonunu azalttı ve bunun tersi de geçerli (Şekil 6g , h). S100A8 inhibitörü paquinimodun eklenmesi, nötrofillerde miR-24-3p inhibisyonunun neden olduğu kolajen-1’deki artışı inhibe etti (Şekil 6i ). Dahası, pankreas yıldız hücrelerine S100A8 eklenmesi, fibrozis belirteçlerinin ekspresyonunu doza bağlı bir şekilde artırdı (Şekil 6j ). Ortak kültür sisteminde, CP hastalarından alınan nötrofillerle birlikte kültürlenen HPSC’lerdeki kolajen-1 protein düzeyi, sağlıklı kontrollerden alınan HPSC’lerdekinden önemli ölçüde daha yüksekti (Ek Şekil S6h ). Paquinimod eklenmesi, CP hastalarından alınan nötrofillerle birlikte kültürlenen HPSC’lerde kolajen-1 protein düzeyindeki artışı engelledi (Ek Şekil S6i ). Daha sonra, benzer etkilerin in vivo olarak meydana gelip gelmediğini test etmek için, bir CP fare modelinde S100A8 inhibisyon tedavisinin anti-fibrotik etkisini araştırdık (Şekil 6k ). Paquinimod uygulaması, CP’nin şiddetini kısmen hafifletti (Şekil 6l ) ancak CP model farelerin pankreas dokularındaki makrofajların M2 polarizasyonu üzerinde önemli bir etkiye sahip değildi (Ek Şekil S6j ). Dahası, paquinimod uygulaması bağışıklık hücrelerinin infiltrasyonunu azalttı ve pankreas nötrofillerinde S100A8 ekspresyonunu da azalttı (Ek Şekil S6k, l ).
Sonra, pankreas yıldız hücrelerinde S100A8’in kesin fonksiyonel reseptörünü tanımlamaya çalıştık. Önceki bir çalışma, S100A8 için üç potansiyel reseptör tanımladı: TLR4, RAGE ve CD36 30 . Bu üç geni başarıyla devre dışı bıraktık (Ek Şekil S6m–o ). HPSC’lerde, Kolajen-1 ve fibronektin (FN) dahil olmak üzere fibrozla ilişkili proteinlerin ifadesi, RAGE veya TLR4’ün devre dışı bırakılmasının ardından S100A8 varlığında inhibe edildi, ancak CD36 devre dışı bırakılmasından sonra inhibe edilmedi (Şekil 6m ). Ayrıca, reaktif oksijen türlerinin (ROS) RAGE veya TLR4 aktivasyonundan sonra yaygın biyolojik süreçlerde yer aldığı bildirildiğinden 31 , 32 , 33 , ROS blokajının pankreas yıldız hücrelerinde S100A8 kaynaklı fibrozu etkileyip etkilemeyeceğini değerlendirdik. Özellikle, NAC (bir ROS inhibitörü) ile tedavi, S100A8 kaynaklı pankreas yıldız hücrelerinde ROS ve fibrozu doz bağımlı bir şekilde inhibe etti (Şekil 6n, o ).
S100A8, makrofajların TLR4 aracılı inflamatuar yanıtı yoluyla β-hücre apoptozunu ve adacık iltihabını şiddetlendirir
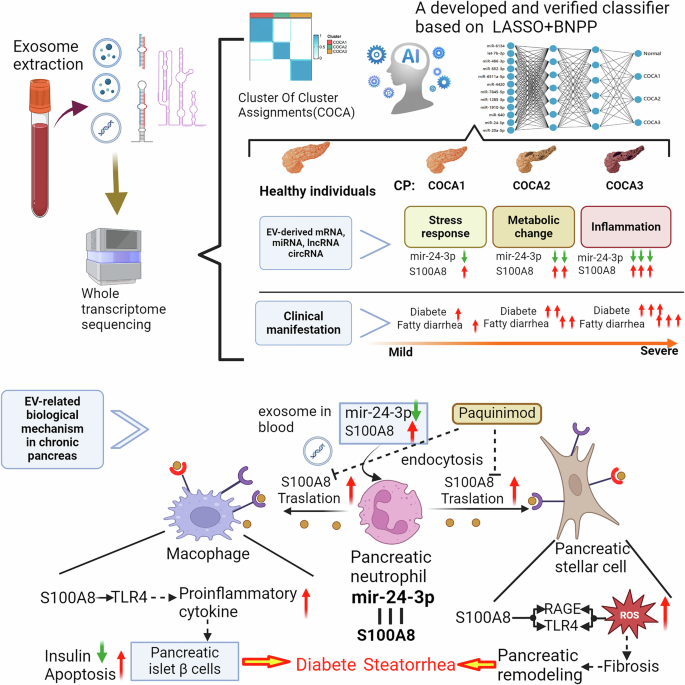
Son olarak, S100A8 aracılı pankreas endokrin bozukluklarının potansiyel mekanizmasını araştırdık. Önceki bir çalışma, S100A8 ve TLR4’ü içeren pankreas β-hücreleri ve makrofajlar arasındaki sinyallemenin tip 2 diyabette β-hücre apoptozunu ve adacık inflamasyonunu şiddetlendirdiğini bildirdi 34 . Benzer bir mekanizmanın, anormal olarak artmış S100A8 seviyelerini CP’deki pankreas β-hücre apoptozu ile ilişkilendirebileceği hipotezini kurduk. Bu fikri test etmek için, RAW264.7 makrofajlarında TLR4’ü baskıladık (Ek Şekil S5j ) ve bunları S100A8 ile veya S100A8 olmadan birlikte kültürledik. WT makrofajlarında, IL-1B, IL-6 ve TNF dahil olmak üzere inflamatuar sitokinlerin mRNA seviyeleri, S100A8 stimülasyonundan sonra arttı ve bu artış TLR4 baskılanmasından sonra tersine döndü (Şekil 7a ). Bu sonuçlar, S100A8’in TLR4 aracılığıyla makrofajların inflamatuar yanıtını desteklediğini göstermektedir. Daha sonra, makrofajların pankreas β-hücrelerinin apoptozu üzerindeki etkisini doğrulamak için bir ortak kültür sistemi tasarladık (Şekil 7b ). Western blot ve immünofloresan, S100A8’in makrofajların varlığında pankreas β-hücrelerinin apoptozunu desteklediğini ortaya koydu. Makrofajlarda TLR4’ün baskılanması, pankreas β-hücrelerinin bu apoptotik fenotipini kısmen tersine çevirebilir ve S100A8 tek başına makrofajların yokluğunda pankreas β-hücrelerinin apoptozunu indükleyemez (Şekil 7c, d ). Bu sonuçlar, S100A8’in makrofajların inflamatuar fenotipini destekleyebileceğini ve S100A8’in makrofajlarda TLR4’ün aktivasyonu yoluyla pankreas β-hücre apoptozunu destekleyebileceğini göstermektedir. Bu çalışmada sunulan önerilen mekanizmayı ve tanı şemasını gösteren diyagramlar Şekil 8’de gösterilmiştir .

Tartışma
CP’nin dünya çapında bildirilen insidansı 100.000’de 42 ila 73 arasında değişmektedir ve zaman içinde artan bir yaygınlık gözlenmektedir 35 . CP’li bireylerin çoğu ayaktan tedavi edilmektedir. Ancak, Amerika Birleşik Devletleri, Birleşik Krallık, Hollanda ve Finlandiya’dan gelen kayıtlar, altı yıllık bir süre içinde bildirilen %30’luk bir artışla yıllık hastane yatışlarında endişe verici bir artış olduğunu göstermektedir ve bu hastalığın büyük sosyoekonomik etkisini vurgulamaktadır. CP ile ilişkili ölüm oranlarının, 6,3-9,8 yıllık ortalama gözlem sürelerine dayanarak %12,8 ile %19,8 arasında olduğu tahmin edilmektedir 35 , 36 , 37 , 38 . Ek olarak, CP’li hastaların yaklaşık %33’ü çalışmaya devam edememektedir 39 . Bu hususlar göz önüne alındığında, araştırmacılar CP’nin şiddetini tahmin etmek için istikrarlı bir sınıflandırma sistemi geliştirmek için önemli çaba sarf etmişlerdir. 1963 Marsilya sınıflandırması morfoloji ve etiyolojiye odaklanmıştı 8 ve daha sonra 1985’te işlevsel korelasyonlara sahip Marsilya-Roma sınıflandırması olarak geliştirildi 9 , 10 . Cambridge sınıflandırması ERP/BT bulgularını kullanarak görüntüleme tabanlı derecelendirmeyi başlattı ancak erken evre işlevsel/histolojik değişiklikleri yansıtmada sınırlamalarla karşılaştı 11 , 12 . Son sistemler şiddeti sınıflandırmak için görüntüleme, semptomlar, müdahaleler ve işlevsel kaybı entegre ediyor 14 , 15 , 16 , 17 , 18 . Ancak, bu sınıflandırmalar moleküler biyobelirteçlere dayanmadığından klinik doğrulukları azalır.
Son yirmi yılda yapılan moleküler çalışmalar, CP heterojenliğini ortaya koymuştur: genetik mutasyonlar tripsin düzenlemesini veya kalsiyum sinyallemesini bozar. Yeni ortaya çıkan moleküler alt tipleme, CP’yi genetik, alkolle ilişkili ve idiyopatik formlar olarak kategorize etmektedir 1 . Ancak, bu etiyolojik çerçevelerde çoklu omik biyobelirteç entegrasyonu eksiktir ve bu da steatore ve diyabet dahil olmak üzere komplikasyon riskinin sınıflandırılmasında hassasiyeti sınırlamaktadır.
Şu anda, CP şiddetinin veya ekzokrin ve endokrin bozuklukların, steatore ve diyabetin neden olduğu iki ciddi komplikasyon riskinin değerlendirilmesi için yaygın olarak kullanılan modeller bulunmamaktadır. Sıvı biyopsi, CP hastalarının pankreas lezyonlarından alınan tek hücreli dizileme verileri ve periferik kan eksozomlarının tüm transkriptom dizilemesini kullanarak, CP hastalarının pankreas hücrelerindeki mRNA değişikliklerinin dolaşımdaki eksozomlarda tekrarlandığını ilk kez gösterdik. Bu bulgu, dolaşımdaki eksozomların pankreasta meydana gelen moleküler değişikliklerin bir yansıması olarak hizmet edebileceğini ve CP ilerlemesini ve komplikasyon riskini izlemek için invaziv olmayan bir yaklaşım önerdiğini göstermektedir. Bu bulgular, prostat kanseri, meme kanseri ve akciğer kanseri dahil olmak üzere farklı kanser türlerini içeren bazı yeni çalışmaların sonuçlarıyla tutarlıdır 25 , 40 , 41 ; özellikle, kan dolaşımındaki eksozom bileşenleri, lokal lezyonlardan kaynaklanan çeşitli kötü huylu fenotiplerle ilişkilendirilmiştir ve yeni biyobelirteçler olarak hizmet edebilir. Mevcut bulgularımız bu fikri kanserden kanser dışı kronik hastalıklara kadar genişletiyor.
Çalışmamızın bir diğer önemli noktası, dolaşımdaki ekzosomların tüm transkriptom dizilemesine dayanan COCA tabanlı bir alt tipleme sisteminin başarılı bir şekilde geliştirilmesi ve kullanılmasıdır. mRNA, miRNA, circRNA ve lncRNA verileri de dahil olmak üzere bu verilerin çok boyutluluğu göz önüne alındığında, uygulanabilir bir çok boyutlu kümeleme algoritmasına ihtiyaç duyulmaktadır. Akciğer kanseri 42 , özofagus kanseri 43 , ürotelyal karsinom 44 ve pankanser veri kümelerine 27 dayalı olanlar da dahil olmak üzere , önceki çok platformlu moleküler kanser alt tipleme çalışmalarının çoğu COCA algoritmasını kullanmıştır. Ancak, bildiğimiz kadarıyla, bu algoritma daha önce kanser dışı kronik bir hastalığa uygulanmamıştır. Çalışmamızda, COCA uygulaması CP alt tipleri olarak tanımlanan 3 küme (COCA1–3) üretti. Her küme, WGCNA ile tanımlandığı gibi, COCA1 için stres yanıtı, COCA2 için metabolik değişiklikler ve COCA3 için inflamasyon olmak üzere farklı biyolojik süreçlerin aktivasyonunu sergiledi. Pankreastan türetilen ekzokrin ve endokrin imza genlerinin mRNA seviyeleri de COCA1’den COCA3’e doğru önemli ölçüde artmıştır. Ayrıca, bu bulgularla tutarlı olarak, steatore ve diyabet insidansı COCA1’den COCA3’e doğru artma eğilimindeydi. Bu sonuçlar, mevcut sınıflandırmamızın geçerliliğini ön olarak doğrulamıştır.
Ekzosomlardaki miRNA’lar nispeten kararlıdır ve bu durum hastalık teşhisi için güçlü biyobelirteçler olarak potansiyellerini vurgular 28 . Mevcut sınıflandırma sistemimizin klinik çeviri potansiyelini artırmak için LASSO + BPNN yoluyla dolaşımdaki ekzosomlara dayalı 12-miRNA sınıflandırıcı oluşturduk. Önceki bir çalışma, BPNN’lerin son derece karmaşık doğrusal olmayan gizli sinir katmanlarında bulunan temel özellikleri etkili bir şekilde çıkarabildiğini ve karmaşık ve doğrusal olmayan problemleri geleneksel lojistik regresyon modellerinden daha kolay ele alabildiğini gösterdi 45 . Mevcut çalışmamızda, COCA1–3 alt tipleri ve sağlıklı örnekler başarıyla ayırt edildi ve bu 12 miRNA için hem dizileme verilerinin yeniden analizinde hem de kaydedilen örnekler arasında qPCR verilerinin analizinde %80’den fazla doğruluk oranı elde edildi. Dahası, modelin doğrulama grubundan gelen qPCR verilerine uygulanması, CP hastalarını sağlıklı bireylerden ayırt etmede güçlü bir yetenek ve COCA1’den COCA3’e doğru steatore ve diyabetin artma eğilimi olduğunu ortaya koydu. Bu sınıflandırma sistemi, CP komplikasyonları için kişiselleştirilmiş risk değerlendirmesine olanak tanır. Örneğin, yüksek riskli olarak tanımlanan hastalar, pankreas fonksiyonunun daha etkin bir şekilde korunması için ekstrakorporeal şok dalgası litotripsi ve/veya endoskopik retrograd kolanjiyopankreatografi gibi erken müdahalelerden faydalanabilir. Tersine, düşük riskli hastalar gereksiz tedavilerden kaçınarak kaynak dağılımını optimize edebilir. Gelecekteki çalışmalar, ekzosom kaynaklı RNA imzalarına dayalı tedavilerin uyarlanmasının klinik sonuçları iyileştirip iyileştirmediğini araştırmalıdır. Ayrıca, bu sınıflandırıcı, herhangi bir klinisyen veya araştırmacının erişebilmesi için Gitee veritabanına yüklenmiştir.
Güncel çalışmamız ayrıca, ilk kez, azalmış eksozom kökenli miR-24-3p ve artmış nötrofil kökenli S100A8’in pankreas fibrozisinde ve pankreas β-hücrelerinin apoptozunda önemli bir rol oynadığını ortaya koydu. Birkaç önceki çalışma, miR-24-3p’nin fibrozisi ve inflamasyonu inhibe ettiğini bildirmiştir 46 , 47 . Burada, azalmış eksozom kökenli miR-24-3p’nin pankreas fibrozisindeki işlevini ve pankreas β-hücrelerinin apoptozunu, nötrofillerdeki S100A8’i doğrudan hedef alarak önlediğini bildirdik. Bir S100 proteini olan S100A8’in, diyabetik nefropati 48 , hepatik fibroz 49 , 50 ve pulmoner fibroz 51 dahil olmak üzere birçok hastalıkta fibroza önemli bir katkıda bulunduğu bildirilmiştir . Ek olarak, önceki bir çalışma S100A8’in pankreas β-hücrelerinde apoptozu teşvik edebileceğini ortaya koymuştur 34 . Bu önceki bulguları genişleterek, S100A8’in pankreas yıldız hücrelerinde TLR4/RAGE ve ROS aktivasyonu yoluyla pankreas fibrozunu teşvik ettiğini ve S100A8’in CP’deki pankreas β-hücreleri üzerindeki proapoptotik etkisinin pankreas makrofaj inflamasyonunu teşvik ederek meydana geldiğini gösterdik. Bu sonuçlar, S100A8 inhibisyonunun steatore ve diyabet dahil olmak üzere CP ile ilişkili hem ekzokrin hem de endokrin bozukluklar için potansiyel bir tedavi stratejisi olabileceğini göstermektedir.
Ancak bu çalışmanın da birkaç sınırlaması vardır. İlk olarak, incelenen tüm örnekler farklı hastanelerdeki örnek depolarından elde edilmiştir. CP hastalarında steatore ve diyabeti tahmin etme yeteneğini doğrulamak için COCA sınıflandırıcımızın daha fazla prospektif çalışmasına ihtiyaç vardır. İkinci olarak, kayıtlı hastalarımızda CP etiyolojisi açıkça tanımlanmamıştır, çünkü TIGAR-O Risk/Etiyoloji Kontrol Listesi 52 Çin hastanelerinde yaygın olarak kullanılmamaktadır. Üçüncü olarak, S100A8’in pankreas β-hücrelerinin apoptozu üzerindeki etkisi in vivo olarak iyi çalışılmamıştır. Ek olarak, paquinimod farelerde CP şiddetini azaltırken, klinik çevirisi dikkatli olmayı gerektirmektedir. Hedef dışı etkileri ve sınırlı doku özgüllüğü uygulanabilirliğini engelleyebilir. Bu sınırlamaları ele almak için daha derinlemesine çalışmalara ihtiyaç vardır.
Sonuç olarak, CP hastalarını sağlıklı bireylerden ayırt etmek için yeni bir 3 aşamalı sistem önerdik ve ekzosomla ilişkili sıvı biyopsi verileri aracılığıyla CP hastalarında steatore ve diyabet riskini tahmin etmek için çevrimiçi bir araç geliştirdik. Ek olarak, çalışmamız ekzosom kaynaklı miR-24-3p’nin nötrofillerde S100A8’i doğrudan hedeflemedeki önemli rolünü ortaya koyarak, nötrofil kaynaklı S100A8’in pankreas yıldız hücrelerinde TLR4/RAGE ve ROS aktivasyonu yoluyla pankreas fibrozunu teşvik ettiğini ve CP’de pankreas β hücrelerinde nötrofil kaynaklı S100A8’in proapoptotik etkisinin pankreas makrofaj inflamasyonunu teşvik ederek indüklendiğini gösterdi.
Malzemeler ve yöntemler
Hastalar ve örnekler
Ekim 2017 ile Ekim 2019 arasında, 140 kronik pankreatit hastası ve 27 sağlıklı donörden plazma örnekleri toplandı ve Çin, Şanghay’daki Changhai Hastanesi’nde donduruldu. Bu kan örnekleri, ERCP kullanılarak planlı pankreas kanalı taşı çıkarılması için hastaneye yatırılan kronik pankreatit hastalarından elde edildi. Pankreas taşları için ERCP’nin daha önce bildirilen preoperatif endikasyonları ve kontrendikasyonlarına dayanarak, akut pankreatit alevlenmesi cerrahi için kontrendikedir ve akut kronik pankreatit atağı geçiren hastalar hastaneye yatırılmaktan hariç tutulmuştur 53 . Ayrıca, Ekim 2018 ile Ekim 2020 arasında, 39 kronik pankreatit hastası ve 5 sağlıklı donörden plazma örnekleri Çin, Zhengzhou’daki Zhengzhou Üniversitesi Birinci Bağlı Hastanesi’nde toplandı ve donduruldu. Bu kronik pankreatit hastalarından kan örnekleri, bu hastalar rutin olarak muayene için yatırıldıklarında toplandı ve akut kronik pankreatit atağı geçiren hastalar hariç tutuldu. Kronik pankreatitin tanısı ve klinik evresi, Çin’de Kronik Pankreatit Tanı ve Tedavi Kılavuzu’na (2018 baskısı) göre takip edildi 54. Bu çalışmaya kaydedilen tüm hastalarımız 20 yaşın üzerinde ve ailelerinde pankreas hastalığı öyküsü yoktu, bu nedenle kalıtsal faktörler dikkate alınmadı. Kan örnekleri sabah EDTA içeren tüpler kullanılarak alındı ve 10 dakika boyunca 4 °C’de 2890 × g’de santrifüj edildi , ardından izole edilen plazma -80 °C’de saklandı. Kronik pankreatit için tüm tanı ve tedavi yöntemleri mevcut kılavuzlara uygun olarak gerçekleştirildi 55 , 56 , 57. Sigara ve alkol tüketimi, demografik veriler (yaş, cinsiyet vb.) ve diabetes mellitus dahil bilgiler Tablo 1 ve 3’te ayrıntılı olarak belgelendi. Tüm hastalardan katılımdan önce yazılı bilgilendirilmiş onam alındı. Bu araştırma için etik onay, Changhai Hastanesi Etik Komitesi (CHEC2017-232) ve Zhengzhou Üniversitesi Birinci Bağlı Hastanesi Etik Komitesi (2018-KY-0324) tarafından verilmiştir. Çalışmaya katılan tüm hastalar, yeni başlayan steatore veya diabetes mellitus tanısı konup konulamayacağını araştırmak için yaklaşık 4 yıl boyunca takip edilmiştir. Bu çalışma, Helsinki Deklarasyonu’nun etik yönergelerine uygundur.
Plazma ekzosomlarının RNA dizilimi
Her numuneden toplam ekzozomal RNA, üreticinin talimatlarına göre TRIzol reaktifi (Invitrogen, ABD) ile ekstrakte edildi. Ekstrakte edilen RNA’ların kalitesi ve saflığı, Agilent 2100 Bioanalyzer (Agilent Technologies, ABD) kullanılarak ölçüldü. Dizileme kütüphanesi, NEXTflex ® Small RNA-Seq Kit v3 (NOVA-5132-05, Bio Scientific Corporation, ABD) veya Ovation Human FFPE RNA-Seq Kütüphane Sistemi (0340-32, NUGEN, ABD) kullanılarak gerçekleştirildi ve her toplam RNA’dan 20 ng’lık bir örnek girdisi kullanıldı. Son olarak, dizileme kütüphanesinin ekspresyonunu HiSeq 2500 (Illumina, Inc., San Diego, CA, ABD) kullanarak profilledik. Düşük kaliteli okumaları kaldırmak ve yüksek kaliteli okumalar elde etmek için Cutadapt yazılımı kullanıldı 58 . Karşılaştırma yazılımı Tophat ( http://ccb.jhu.edu/software/tophat/index.shtml ), temiz okumaları bir gen konumuyla açıklanan bir referans genomuna eşlemek için kullanıldı ve Bowtie2 ( http://ccb.jhu.edu/software/tophat/index.shtml ) referans genomunun indeksini oluşturmak için uygulandı. CircRNA’lar ve circRNA’ların konak geni find_circ 59 ve CIRCexplorer 60 tarafından tanımlandı . Transkriptleri birleştirmek için StringTie yazılımını kullandık ve bilinen lncRNA’ları taramak için Perl betiklerinden yararlandık. LncRNA’ların ve mRNA’ların kantitatif analizi Ballgown R paketi kullanılarak gerçekleştirildi.
Diferansiyel ifade
İki grubun farklı ekzomal mRNA, lncRNA, miRNA ve circRNA ekspresyon analizleri R’de limma paketi kullanılarak gerçekleştirildi. Katlama değişimi (FC), CP grubu ile kontrol grubu arasındaki farklı ekspresyonun bir göstergesi olarak kabul edildi. Farklılıkların istatistiksel anlamlılığını değerlendirmek için t -testleri kullanıldı. Farklı ekspresyonu göstermek için p -değerleri < 0,05 olarak kabul edildi.
LncRNA’nın hedef genlerinin tahmini
LncRNA hedef gen tahmini LncRNA’ların hedef gen tahmini iki şekilde gerçekleştirildi: cis etkili hedef gen tahmini ve trans etkili hedef gen tahmini. Cis etkili düzenleyici eleman teorisine dayanarak , lncRNA’dan 10 kb uzaklıkta bulunan protein kodlayan genler potansiyel cis etkili hedef olarak seçildi 61. Trans etkili hedef tahmini için ise , kodlayan genler ve lncRNA’lar arasındaki Pearson korelasyon katsayıları hesaplandı ve trans etkili düzenleyici elemanların tanımlanması için analiz edildi (Katsayılar > 0,5).
miRNA-mRNA ile ilgili etkileşim çiftlerinin tanımlanması
Deneysel Kanıtlara sahip 68.176 potansiyel miRNA-mRNA etkileşim ağı miRTarBase’den indirildi (Ek Tablo S9 ). Ardından, 68.176 tahmin edilen miRNA-mRNA düzenleyici ilişkisi, miRNA-DEM düzenleyici ilişkisini elde etmek için DEM verileriyle entegre edildi. Ardından, ceRNA ağları Cytoscape (sürüm 3.6.1; http://www.cytoscape.org/ ) kullanılarak geliştirildi .
Yol zenginleştirme analizi
Farklı şekilde ifade edilen miRNA’lar ( cis etkili düzenleyici ve trans etkili düzenleyici lncRNA’ların mRNA’ları) tarafından hedeflenen mRNA’lar , circRNA’nın konak genleri tarafından transkribe edilen mRNA’ları, clusterProfiler paketi kullanılarak GO ve Kyoto Encyclopedia of Genes and Genomes yol zenginleştirme analizine tabi tutuldu. Bu analiz, zenginleştirilmiş biyolojik süreçleri, moleküler işlevleri, hücresel bileşenleri ve bu mRNA’larla ilişkili yolları belirlemek için gerçekleştirildi. Zenginleştirme sonuçları için Benjamini-Hochberg ayarlı P değerinin (FDR) 0,05’ten düşük olması anlamlı kabul edildi.
scRNA-seq veri işleme, küme açıklaması ve insan pankreası için veri entegrasyonu
13 kronik pankreas dokusunun tek hücre verileri http://singlecell.charite.de/cellbrowser/pancreas/ ?ds=Chronic_Pancreatitis 26 adresinden Seurat paketi (v4.4.0) 62 kullanılarak indirildi . 500’den az gen ve 5000’den fazla gen ifade eden hücreler, %10’dan fazla mitokondri oranına sahip hücreler, toplam mRNA molekülü sayısı 500’den az tespit edilen hücreler, eritrosit içeriği %3’ten fazla olan hücreler ve üçten az hücrede ifade edilen genler, aşağı akış analizinden hariç tutuldu. Ek hücre filtrelemesi için SoupX paketinden (v1.6.2) 63 “SoupX” yöntemi ve DoubletFinder paketinden (v2.0.3) 64 “doubletFinder_v3” yöntemi uygulandı. Filtrelenen veriler daha sonra log-normalize edildi ve ölçeklendirildi; UMI sayımları ve mitokondriyal okuma yüzdesine bağlı hücre-hücre değişimi geriletildi. Çapalama için toplam 2000 özellik (“FindVariableFeatures” işlevi) ve hizalama için 40 boyut (“Integrate Data”) kullanıldı. Hücre kümelemesi, 0,1 çözünürlükte “FindClusters” işlevi ile gerçekleştirildi. Boyut indirgemesi, “Run UMAP” işlevi ile gerçekleştirildi ve UMAP ile görselleştirildi. Alt grup hücre kümelemesi için, farklı türdeki hücreler ayrı ayrı çıkarıldı ve görsel incelemeye dayalı olarak farklı çözünürlükler kullanılarak ilgili ilk 30 temel bileşenine göre kümelendi.
Entegre COCA
Dört platformdan her birinden türetilen alt tipler, mRNA, miRNA, lncRNA ve circRNA, yukarıda açıklandığı gibi hesaplandı. Dört platformun her biri için alt tip çağrıları, Monti ve ark. 65’teki tanımlara uygun olarak her alt tip için bir dizi gösterge değişkenine kodlandı. İkili matris, ConsensusClusterPlus R paketinde 66 konsensüs kümelemesi için girdi olarak kullanıldı. Özellikle, Pearson korelasyon mesafesi metriğine dayalı PAM yöntemi kullanılarak 2 ila 10 kümeden %80 yeniden örneklemenin 1000 yinelemesi yapıldı. İstenen küme sayısı , CDF altındaki alandaki oransal artış Δ( K ) ‘ye göre belirlendi.
ve A( K ), K kümelerinin ( K = 2, 3, …, 10) eğri altında kalan alanıdır . Sayıdaki daha fazla artışın A( K )’yi önemli ölçüde artırmadığı ve ana grupların fikir birliği kümeleme haritasında sabit kaldığı küme sayısını seçtik.
Ekzokrin ve endokrin imza genlerinin tanımlanması ve alt gruplandırılması
Her hücre alt kümesi için spesifik genleri tanımlamak amacıyla Seurat’taki “FindAllMarkers” fonksiyonunu uyguladık. Her hücre kümesi/alt kümesine özgü belirteç genlerinin seçimi için, iki grup (bir hücre kümesi/alt kümesi ile diğer hücreler) arasındaki log 2 FC’yi Wilcoxon sıralama toplamı testi (varsayılan parametreler) ile “findallmarkers” fonksiyonunu kullanarak hesapladık. Hücre tiplerinin ve her hücre alt grubunun gen belirteçleri önceki bir çalışmaya göre seçildi ve hücre alt grubu bu gen belirteçlerine göre açıklandı. Ardından, ekzokrin bozukluk imza genleri, Acinar REG + hücrelerinde ve aktive edilmiş yıldız hücrelerinde ilk 40 gen (log 2 FC ile hesaplanan) olarak tanımlandı (Ek Tablo S6 ) ve endokrin imza genleri de tanımlandı; bunlar alfa hücrelerinin ve beta hücrelerinin ilk 40 belirteç geninden (log 2 FC ile hesaplanan) oluşuyordu ( Ek Tablo S7 ). Koekspresyona tabi tutulan ekzokrin ( X ekseni) imza genlerinin ve endokrin ( Y ekseni) imza genlerinin medyan ifade düzeyleri, her bir numunenin koordinat grafiklerindeki dağılımını görselleştirmek için kullanıldı.
WGCNA
Her COCA kümesiyle ilişkili yüksek korelasyonlu gen kümeleri , R (v 3.6.3) kullanılarak yürütülen bir WGCNA yaklaşımı 67 aracılığıyla tanımlandı . Gen modülleri arasındaki güçlü korelasyonları daha iyi tespit etmek için pickSoftThreshold R fonksiyonu ile yumuşak eşikleme gücü ( β ) belirlendi. Daha sonra modülleri tespit etmek için hiyerarşik kümeleme analizleri gerçekleştirildi. Etkileşim gücü, ilişkileri değerlendirmek için gen önemi (GS) ve modül üyeliği (MM) hesaplanarak Heatmap araçları paketiyle değerlendirildi.
COCA sınıflandırması ve CP tanısı için bir sınıflandırıcı oluşturulması
Araştırma bulgularımızın klinik olarak uygulanabilirliğini kolaylaştırmak için, kronik pankreatit ve alt tiplerini doğru bir şekilde tahmin etmeyi amaçlayan, miRNA ekspresyon profillerini modellemek için esnek geri yayılım tabanlı bir sinir ağı algoritması kullandık ve böylece çalışmamızın klinik uygulanabilirliğini artırdık. İş akışı aşağıdaki gibidir:
- 1.Özellik Seçimi: Her alt tip ve sağlıklı örneklem için, öncelikle tek değişkenli lojistik regresyondan P değeri < 0,01 olan mikroRNA’ları tuttuk. Ardından, tüm örneklerden verilerin %70’ini rastgele örneklemek için Bootstrapping yöntemini kullandık ve 1000 yineleme üzerinden lojistik regresyon gerçekleştirdik. Yeniden örnekleme süreçlerinin %95’inden fazlasında anlamlı kalan genleri ( P < 0,05) tuttuk. Bunu takiben, boyut azaltma ve model sadeleştirme için Lasso algoritmasını kullandık ve sıfır olmayan Lasso katsayılarına sahip bu özellikleri modelleme için girdi değişkenleri olarak tuttuk.
- 2.Hiperparametre Optimizasyonu: Tüm örnekleri %80:20 oranında eğitim ve test kümelerine ayırdık. Sinir ağı modeli, R yazılımındaki “neuralnet” paketi kullanılarak oluşturuldu. Optimize edilecek parametreler arasında öğrenme hızı, kayıp fonksiyonu, aktivasyon fonksiyonu, gizli katman sayısı ve her katmandaki düğüm sayısı yer alıyordu. Hiperparametre optimizasyonu, ızgara arama yaklaşımı kullanılarak gerçekleştirildi ve nihai modelin parametreleri, test kümesinde en yüksek doğruluğu sağlayan kombinasyona göre seçildi.
- 3.Model Doğrulaması: Model performansı, eğitim seti, test seti ve tüm veri seti üzerinde karışıklık matrisleri kullanılarak değerlendirildi.
12 miRNA COCA sınıflandırıcısında temel miRNA-mRNA etkileşim çiftini belirleyin
Prosedür akış şeması Şekil 5a’da gösterilmiştir. İlk olarak, daha önce edinilen Ek Tablo S9’dan potansiyel bağlanma yeteneğine sahip 12-miRNA ile ilişkili mRNA taranır . Bu prosedürde 2750 miRNA-mRNA tanımladık (Ek Tablo S14 ). Daha sonra, tüm transkriptom dizilemesi sonuçlarımıza dayanarak, kontrol grubu, COCA1 , COCA2 ve COCA3 arasında FDR < 0,05 ve R < -0,3 kriterlerini karşılayan korelasyon katsayılarına ( R değeri, Pearson korelasyon katsayısı) sahip 31 miRNA-mRNA çifti tanımlandı (Ek Tablo S15 ). Daha sonra, tutarlı eğilimlere sahip miRNA’lar ( normal grup, COCA1-3 grupları dahil olmak üzere bitişik gruplar arasındaki çiftler halinde en az üç karşılaştırma için P < 0,05) tarandı. Son olarak, mRNA taranan miRNA ile etkileşime girdi ve tutarlı eğilimler ( normal grup, COCA1–3 grupları dahil olmak üzere komşu gruplar arasındaki en az üç çift karşılaştırmada P < 0,05) tanımlandı (S100A8 ve miR-24-3p).
Hücre kültürü
HL-60 hücre hattı (RCB3683, RIKEN BioResource Center, Ibaraki, Japonya) %10 (v/v) FBS ve %1 penisilin/streptomisin (PS) içeren bir RPMI 1640 besiyerinde kültürlendi. HL-60 hücrelerini nötrofil benzeri hücrelere farklılaştırmak için hücreler 3 gün boyunca %1,25’lik DMSO ile kültürlendi. HPSC’ler, Houston, Teksas, ABD’deki Anderson Kanser Merkezi’nin Kanser Biyolojisi Bölümü’nden Prof. Logsdon CD tarafından cömertçe sağlandı 68 . Ham 264.7 hücreleri Amerikan Tip Kültür Koleksiyonu’ndan (ATCC, ABD) alındı. HEK-293 hücreleri de Amerikan Tip Kültür Koleksiyonu’ndan alındı. Hem HPSC’ler, HEK-293 T hücreleri hem de Ham 264.7 hücreleri %10 FBS ve %1 PS ile desteklenmiş DMEM’de kültürlendi. Adacık β hücresi MIN6 hücreleri (ATCC, ABD), %1 PS, %10 FBS ve %1 β-merkaptoetanol içeren DMEM’de (25 mmol/L glikoz) kültüre alındı. Tüm hücreler, 37 °C’de %5 CO2 ortamında, her 2 günde bir ortam değiştirilerek kültüre alındı .
Hücre transfeksiyonu
Hücreleri ileri analizler için üstel büyüme aşamasında topladık. Özellikle, HL-60 hücreleri 3 gün boyunca %1,25 DMSO ile muamele edildi. hsa-miR-24-3p taklitleri, hsa-miR-24-3p inhibitörü, NC taklidi ve NC inhibitörü (Hanbio, Şanghay, Çin), üreticinin talimatları doğrultusunda Lipofectamine 2000 (Thermo Fisher Scientific, ABD) kullanılarak ADSC’lere transfekte edildi. HPSC’ler için, CD36’yı baskılamak için küçük girişimci RNA (siRNA) (si-36), RAGE siRNA (si-RAGE), TLR4 siRNA (si-TLR4) ve negatif kontrol NC (si-NC), RiboBio (Guangzhou, Çin) tarafından tasarlanıp sentezlendi. RAW264.7 hücresi için TLR4 siRNA (si-TLR4) ve negatif kontrol NC (si-NC) de RiboBio (Guangzhou, Çin) tarafından tasarlandı ve sentezlendi. HPSC’ler ve RAW264.7, Lipofectamine 2000 (Thermo Fisher Scientific, ABD) kullanılarak siRNA’larla transfekte edildi. Hücreler transfeksiyondan yaklaşık 12 saat önce tamamlanmış ortama ekildi. Daha sonra, Lipofectamine 2000 ile karıştırılmış siRNA’lar, taze Opti-MEM ortamı (Gibco, ABD) ile hücrelere eklendi. SiRNA’lar 50 nM konsantrasyonda transfekte edildi. 6 saat sonra, siRNA’ları ve Lipofectamine 2000’i içeren ortam tamamlanmış ortamla değiştirildi. HPSC hücrelerini uyarmak için farklı seviyelerde S100A8 proteini (HY-P70531, MCE şirketi, ABD) kullanıldı.
Nötrofil-pankreatik yıldız hücre ortak kültür testi
Nötrofil-pankreas yıldız hücresi ko-kültür sistemi için şematik diyagramlar Şekil 7F’de gösterilmiştir. 6 kuyulu bir transkuyu sisteminde, HPSC’ler (1 × 10 6 ) alt hazneye plaka edildi, DMSO indüksiyonundan sonra farklı HL-60 hücre grupları (1 × 10 6 ) ise üst hazneye eklendi; gözenek boyutu 0,4 μm idi. Tüm hücreler %10 FBS, 50 U/mL penisilin (Gibco, ABD) ve 50 μg/mL streptomisin (Gibco, ABD) ile desteklenmiş RPMI 1640’ta büyütüldü. 2 saatlik ko-kültürden sonra, ko-kültür sisteminin iki grubuna 10 μg/mL dozunda paquinimod (HY-100442, MCE company, ABD) eklendi. Daha sonra HPSC’lerin her bir grubu 48 saat sonra alt bölmeden hasat edildi.
Makrofaj-adacık hücresi ortak kültür testi
Makrofaj-adacık hücre ko-kültür sistemi için şematik diyagramlar Şekil 7B’de gösterilmiştir . Altı kuyulu bir transkuyu sisteminde, mini6 hücreleri (1 × 10 6 ) alt hazneye yerleştirilirken, farklı RAW264.7 grupları üst hazneye eklendi; gözenek boyutu 0,4 μm idi. Tüm hücreler, %1 PS, %10 FBS ve %1 β-merkaptoetanol ile desteklenmiş DMEM’de (25 mmol/L glikoz) büyütüldü. 2 saatlik ko-kültürden sonra, ko-kültür sisteminin iki grubuna 10 μg/mL dozunda paquinimod (HY-100442, MCE company, ABD) eklendi. Daha sonra, mini6 hücrelerinin her bir grubu 48 saat sonra alt hazneden toplandı.
Veri kaynağı ve tanı değerinin belirlenmesi
15 osteoartrit hastasının ve 3 romatoid artrit hastasının kan ekzozomal miRNA ekspresyonu sırasıyla GSE185059 ve GSE218599’dan indirildi . İki GEO veri setinin ve yerel kohortumuzun toplu etki etkisini ortadan kaldırmak için R yazılımı 69’da SVA paketi kullanıldı . Ardından, miRNA değeri COCA algoritmasına girildi ve tanı değeri hesaplandı.
scRNA-seq veri işleme, küme açıklaması ve fare pankreası için veri entegrasyonu, ekzosom salgılama aktivite indeksi çıkarımı
Üreticinin tanıtımına göre, scRNA-seq kütüphaneleri Tek Hücreli 5′ Kütüphane ve Jel Boncuk Kiti kullanılarak oluşturuldu. Kütüphaneler, hücre başına en az 77.618 okuma dizileme derinliğine sahip bir Illumina Novaseq 6000 dizileyici kullanılarak, çift uç 150 bp okuma stratejisiyle (Çin’deki Oyi şirketi tarafından gerçekleştirildi) dizilendi. Kütüphaneleri hazırlamak için üreticinin protokolüne göre Chromium Tek Hücreli 3′ Reaktif v3 kitleri kullanıldı. Tek hücreli süspansiyonlar, emülsiyonlarda tek hücreli jel boncukları (GEM’ler) oluşturmak için Chromium Tek Hücre Kontrol Cihazına (10X Genomics, Pleasanton, CA, ABD) yüklendi. GEM’lerin oluşturulmasından sonra, ters transkripsiyon reaksiyonları gerçekleştirildi. Ardından, cDNA çoğaltıldı, parçalandı, uç onarımı yapıldı, A-kuyruklu hale getirildi, indeks adaptörü bağlandı ve kütüphane çoğaltmasına tabi tutuldu. Her kütüphane bir NovaSeq 6000 platformunda (Illumina, San Diego, CA, ABD) dizilendi ve 150 bp’lik eşleştirilmiş uç okumaları üretildi. 10X Genomics tarafından sağlanan Cell Ranger yazılım hattı (sürüm 3.1.0), hücresel barkodları ayırmak, STAR hizalayıcıyı kullanarak okumaları genom ve transkriptoma eşlemek ve örnekler arasında normalleştirilmiş toplam verileri üretmek için gerektiği gibi okumaları aşağı örneklemek için kullanıldı ve gen sayımlarının hücrelere göre bir matrisi üretildi 70 . Ardından, her vaka için üç dosya (barkodlar, özellikler ve matris) üretildi. Üç dosya, Python 3.9’daki EVtrans algoritması 71 ile işlendi . “EVtras.sEV_recognizer()” ve “SEVtras.ESAI_calculator()” ile h5ad formatında ekzosomla ilgili matris üretildi. Aynı zamanda, matris Scanpy (sürüm 1.9.1) 72 kullanılarak analiz edildi .
Her vaka için üç dosyayı (barkodlar, özellikler ve matris) indirdik ve bunları Scanpy 72 nesnesini oluşturmak için kullandık . Bir veri setinde (GSE159977) kullanılan bağışıklık hücreleri göz önüne alındığında, daha ileri analizler için yalnızca bağışıklık hücrelerini çıkardık. Bir örnekte üçten az hücrede ifade edilen genler ve 200’den az gen ifade eden hücreler hariç tutuldu. Sayım matrisinde ifade edilen gen sayısına ve mitokondriyal gen sayımlarının yüzdesine göre hücrelerde daha ileri kalite kontrolü gerçekleştirildi. >1000 RNA transkripti ve <25.000 RNA transkripti ve <%15 mitokondriyal gen sayımına sahip hücreler filtrelendi. >200 gen ve <55.000 gen içeren hücreler bir sonraki adıma filtrelendi. Daha sonra Scanpy’deki “scanpy.pp.normalize_total” işlevini kullanarak veri matrisini normalleştirmek için kütüphane boyutu düzeltme yöntemini uyguladık. Sonraki analiz için logaritmik normalleştirilmiş bir veri matrisi kullanıldı.
Boyut azaltma ve gözetimsiz kümeleme
Boyut indirgeme ve denetimsiz kümeleme, Scanpy’deki iş akışına göre gerçekleştirildi. “highly_variable_nbatches ≥ 4” parametresiyle “scanpy.pp.highly_variable_gene” işlevini kullanarak akış analizi için oldukça değişken genleri seçtik. Toplam 2303 oldukça değişken gen belirlendi. Hücre döngüsünün etkisini araştırmak için “sc.tl.score_genes_cell_cycle” işlevini kullanarak “S_score” ve “G2M_score” değerlerini hesapladık. Daha sonra, hücre başına toplam sayımların, ifade edilen mitokondriyal genlerin yüzdesinin, “S_score” ve “G2M_score” değerlerinin etkileri “scanpy.pp.regress_out” işlevi kullanılarak regresyona uğratıldı. Ayrıca her geni “max_value = 10” parametresiyle “scanpy.pp.scale” kullanarak birim varyansa ölçekledik. Veri ön işlemesinden sonra, bir PCA gerçekleştirerek verilerin boyutunu azalttık. Varyasyonun ana eksenlerini ortaya çıkarmak ve verileri “svd_solver = “arpack”, n_pcs = 11” parametreli “scanpy.tl.pca” fonksiyonu ile gürültüden arındırmak için bir PCA matrisi hesaplandı. Ayrıca, birleştirilmiş veri kümelerinin boyutluluğu “scanpy.tl.umap” fonksiyonu tarafından uygulanan UMAP kullanılarak azaltıldı. Sonra, hücrelerin mahalle grafiğini kümelemek için Leiden grafik kümeleme yöntemini kullandık. Her kümenin belirteç genleri “scanpy.tl.rank_genes_groups” fonksiyonu kullanılarak belirlendi. Her kümenin belirteç genleri önceki tek hücreli çalışmalardan belirlendi, özellikle C1qa ve Mrc1 makrofajları tanımlamak için, Nkg7, Cd3d, Gata3 ve Il7r T veya NK hücrelerini tanımlamak için, Prss2 ve Cpa1 Acinar hücrelerini tanımlamak için kullanıldı; Sdc4, Adamtsl1, Col1a1 ve Col6a1 Fibroblastı tanımlamak için kullanıldı; Nötrofilleri tanımlamak için S100A9 ve S100A8; moDC’yi tanımlamak için Plbd1 ve Cd83; Naif B hücrelerini tanımlamak için Ighd ve Ms4a1; Plazmayı tanımlamak için Jchain; Kırmızı hücreleri tanımlamak için Hbb-bs kullanıldı. Ardından, ekzozom verileri ve tek hücre verilerini birleştirmek için sEVtrans kullanıldı ve her hücre tipinin ekzozom salgılama aktivitesi “SEVtras.ESAI_calculator” aracılığıyla hesaplandı. Ardından, veriler h5ad’den Seurat nesnelerine dönüştürüldü. Son olarak, veriler Seurat V5 ve SCP paketi ( https://zhanghao-njmu.github.io/SCP/ ) ile R yazılımı 4.0.2’de (İstatistiksel Hesaplamalar için R Vakfı, Viyana, Avusturya) görselleştirildi.
Şematik çizim ve istatistiksel analizler
Tüm deneysel veri işleme Python 3.9 (Python Yazılım Vakfı, ABD), R yazılımı 4.0.2 (İstatistiksel Hesaplamalar için R Vakfı, Avusturya) ve GraphPad Prism 8.01 yazılımı (GraphPad Şirketi, ABD) kullanılarak gerçekleştirildi. Sonuçlar ortalama ± standart sapma (SS) olarak gösterilmiştir. İki grup arasındaki farkların anlamlılığı t -testi ve tek yönlü varyans analizi ile belirlendi. Anlamlılık, P < 0,05 değerinde kabul edildi . Bu çalışmada sunulan şematik diyagramlar, Biorender ( https://app.biorender.com/ adresinden erişilebilir) kullanılarak oluşturuldu ve kullanımı için gerekli izinler alındı.
Veri kullanılabilirliği
Mevcut çalışmada kullanılan tüm transkriptom dizileme verileri ve fare tek hücre dizileme verileri, CRA018391 ve CRA021519 gönderim numaralarıyla GSA veritabanında ( https://ngdc.cncb.ac.cn/ ) kamuya açıktır. Tek hücre dizileme verileri kamuya açık veritabanlarından ( http://singlecell.charite.de/cellbrowser/pancreas/?ds=Chronic_Pancreatitis ) edinilebilir . Geliştirilen COCA kronik pankreatit sınıflandırıcısı https://gitee.com/haojiehuangSMMU/COCA-classifier-of-chronic-pancreatitis adresinden indirilebilir ve R yazılımıyla kullanılabilir.
